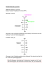195 kB - Johannes Gutenberg
Transcrição
195 kB - Johannes Gutenberg
Johannes Gutenberg-Universität Mainz Institut für Mikrobiologie und Weinforschung FI-Übung: Identifizierung, Wachstum und Regulation (WS 2004/05) Sebastian Lux Datum: 19.1.2005 6. Induktion der Galactosidase von Escherichia coli 1. Theoretische Grundlagen Befinden sich Glucose und Lactose in einer Lösung, so verbraucht E. coli zuerst sämtliche Glucose, bevor er auf Lactosestoffwechsel umschaltet. Verantwortlich für diesen Regelungsmechanismus ist das lac-Operon, welches einer positiven und einer negativen Kontrolle unterliegt. Das lac-Operon besteht aus einem Promotor, welcher Bindungsstelle für das Regulatorprotein CAP („Catabolite Activator Protein“) ist, einem Operator, welcher Bindungsstelle für einen Repressor ist, sowie den drei Strukturgenen Strukturgene codieren für die Enzyme lacZ, lacY und lacA. Die Galactosidase, welche Lactose in Galactose und Glucose spaltet, Permease, welche für den Transport von Lactose in die Zelle verantwortlich ist, sowie für eine Transacetylase. Abb. 1: lac-Operon Als Induktor fungiert das Lactose-Isomer Allolactose. Diese hemmt den lacRepressor, welcher bei Abwesenheit von Lactose an den Operator gebunden ist und so die Transkription verhindert. Indem Allolactose an den Repressor bindet, verändert sich dessen Konfirmation und er kann nicht mehr an den Operator binden, wodurch die Transkription ermöglicht wird. Allerdings bedarf es zusätzlich noch einer positiven Kontrolle. Erst wenn das Regulatorprotein CAP am Promotor bindet, startet die Transkription endgültig. CAP benötigt den Signalstoff cAMP, welcher bei Glucosemangel als Hungersignal gebildet wird. Regulierend für die cAMP-Bildung ist das Phosphotransferasesystem. Ist Glucose vorhanden, so wird diese über Enzym III phosphoryliert, bei Abwesenheit von Glucose induziert die phosphorylierte Form von EIII die Synthese von CAMP durch die Adenylatcyclase. Abb. 2: Phosphotransferasesystem Somit konkurriert Glucose erfolgreich mit Lactose um den Platz als verwendetes Substrat. Dieses als „Katabolit-Repression“ bezeichnete Phänomen ist insofern plausibel, da Glucose für E. coli energetisch günstiger ist. Lactose muss erst gespalten werden und Galactose erst umgewandelt werden, bevor sie dem Hexoseabbau zugeführt wird. Auch andere Kohlenhydrate wie z.B. Mannit werden gegenüber Lactose bevorzugt. Ist die bevorzugte Energiequelle verbraucht, steigt E. coli auf das nächstgünstigere Substrat um. Bei diesem Wechsel der Energiequelle kommt es zu einem kurzzeitigen Wachstumsstillstand, bevor die neue Energiequelle verwertet wird. Dieses Phänomen wird als Diauxie bezeichnet. 2. Material und Methode Vom E. coli-Stamm AN387 werden Vorkulturen auf Lactose, Glucose sowie Lactose+Glucose gezüchtet. Diese werden am Versuchstag in sterile Reagenzgläser mit M9-Medium und der entsprechenden C-Quelle überführt und für zwei Stunden bei 37°C inkubiert. Um die Bestimmung durchführen zu können, wird mit Chloroform und Sodiumdodecylsulfat die Permeabilität der Zellmembranen erhöht. Nachgewiesen wird die und ONP Galactosidase mit Hilfe von ONPG, welches in Galactose Nitrophenol) gespalten wird. Der Gehalt kann photometrisch bestimmt werden und gibt letztendlich Aufschluss über die Aktivität (in Miller-Units) abhängig von den drei Substraten. Von jeder Kultur werden drei Messungen durchgeführt, plus jeweils eine Leermessung nur mit Medium. Gestartet wird die Reaktion durch Zugabe von ONPG, gestoppt wird sie durch Zugabe von Natriumcarbonat, wodurch der pH-Wert steigt und die Galactosidase ihre Funktion verliert. Nachdem die Proben zentrifugiert wurden (um die aufgelösten Zellreste zu entfernen) werden die photometrischen Messungen durchgeführt. Für die Berechnung der Miller-Units müssen neben den Extinktionen auch die optische Dichte der jeweiligen Kultur sowie die Inkubationszeit Versuchsansatzes und das Probevolumen mit einbezogen werden. Für Details siehe Skript. des 3. Ergebnisse Tabelle 1: Messwerte Glucose Lactose Lac + Glc 0,48 0,25 0,55 0,06 0,50 0,26 0,58 0,06 0,49 0,255 0,565 0,06 0,43 0,195 0,505 0 0,057 0,339 0,097 2 0,055 0,384 0,088 3 0,059 0,369 0,092 E420 MW 0,057 0,364 0,092 E420 BW 0,057 0,055 0,058 0 0,309 0,034 30 5 22 0 1585 15 OD578 MW OD (MW-BW) E420 E 1 (MW-BW) t [min] Miller-Units BW Beispielrechnung anhand der Lactosewerte: 1000 * E(MW-BW) Aktivität [Miller-Units] = _____________________________ Zeit [min] * VProbe [ml] * OD (MW-BW) 1000 * 0,309 = ___________________ = 1584,62 à 1585 5 min * 0,2 ml * 0,195 4. Diskussion Der Versuch liefert recht deutliche Ergebnisse. Die Aktivität bei Glucose als Substrat ist erwartungsgemäß gleich null. Glucose wird mit Hilfe des Phosphotransferasesystems phosphoryliert, wodurch die cAMP-Konzentration erniedrigt wird. Infolgedessen bindet der cAMP-CAP-Komplex nicht am Promotor und es findet keine Transkription statt. Zudem bleibt der lac-Repressor bei Abwesenheit von Lactose an den Operator gebunden, was ebenfalls die Galactosidase-Transkription verhindert. Im reinen Lactose-Medium beträgt die Aktivität von Galactosidase 1585 Miller- Units. Glucose wird nicht phosphoryliert, da nicht vorhanden, somit wird vermehrt cAMP gebildet, welches zusammen mit CAP an den Promotor bindet. Desweiteren induziert Allolactose die Transkription von sie an den lac-Repressor bindet und ihn so inaktiviert. Galactosidase, indem Etwa um den Faktor 100 geringer ist die Aktivität, wenn sowohl Lactose als auch Glucose im Medium vorliegen. Zwar wird der lac-Repressor durch Allolactose inaktiviert, doch da Glucose gleichzeitig die Bildung von cAMP hemmt, werden die Strukturgene des lac-Operons nicht transkribiert. Somit entscheidet die positive Kontrolle des lac-Operons darüber, ob eine Transkription stattfindet oder nicht. Womöglich würde man zu einem späteren Zeitpunkt eine höhere Enzymaktivität messen können, da die Bakterien sich evtl. zum Zeitpunkt der Messung noch in der Wachstumsstillstandphase beim Wechsel zwischen den beiden Substraten befinden. Literatur: MADIGAN, MICHAEL T. et. al (2001): Brock. Mikrobiologie, Heidelberg. SCHLEGEL, HANS G. (1992): Allgemeine Mikrobiologie. Stuttgart.