a expressão gênica dos rnas não codificadores hotair, malat1, hulc
Transcrição
a expressão gênica dos rnas não codificadores hotair, malat1, hulc
Vanessa Regina Maciel Uzan A EXPRESSÃO GÊNICA DOS RNAs NÃO CODIFICADORES HOTAIR, MALAT1, HULC E BCYRN1 EM OSTEOSSARCOMA Dissertação apresentada ao Programa de Pós Graduação da Fundação PIO XII – Hospital de Câncer de Barretos para obtenção do Título de Mestre em Ciências da Saúde. Área de concentração: Oncologia Orientador: Prof. Dr. Luiz Fernando Lopes Co-orientador: Dr. Daniel Onofre Vidal Barretos, SP 2015 FICHA CATALOGRÁFICA Preparada por Rafael de Paula Araújo CRB 8/9130 Biblioteca da Fundação Pio XII – Hospital de Câncer de Barretos U99e Uzan, Vanessa Regina Maciel A expressão gênica dos RNAs não codificadores HOTAIR, MALAT1, HULC e BCYRN1 em osteossarcoma / Vanessa Regina Maciel Uzan. - Barretos, SP 2015. 84 f. : il. Orientador: Luiz Fernando Lopes. Dissertação (Mestrado em Ciências da Saúde) – Fundação Pio XII – Hospital de Câncer de Barretos, 2015. 1. Osteossarcoma. 2. RNAs não codificadores longos. 3. Metástase. 4. Expressão Gênica. 5. Reação em Cadeia da Polimerase em Tempo Real. 6. Biomarcadores. I. Autor. II. Lopes, Luiz Fernando CDD 572.86 FOLHA DE APROVAÇÃO Vanessa Regina Maciel Uzan A expressão gênica dos RNAs não codificadores HOTAIR, MALAT1, HULC E BCYRN1 em osteossarcoma Dissertação apresentada ao Programa de Pós-Graduação da Fundação Pio XII – Hospital de Câncer de Barretos para obtenção do Título de Mestre em Ciências da Saúde - Área de Concentração: Oncologia Data da aprovação: 26/02/2015 Banca Examinadora: Prof. Dr. Antônio Sergio Petrilli Instituição: Instituto de Oncologia Pediátrica - Universidade Federal de São Paulo – UNIFESP Prof. Dr. Rui Manuel Vieira Reis Instituição: Fundação Pio XII – Hospital de Câncer de Barretos Prof. Dr. Luiz Fernando Lopes Orientador – Presidente da Banca “Esta dissertação foi elaborada e está apresentada de acordo com as normas da PósGraduação do Hospital de Câncer de Barretos – Fundação Pio XII, baseando-se no Regimento do Programa de Pós-Graduação em Oncologia e no Manual de Apresentação de Dissertações e Teses do Hospital de Câncer de Barretos. Os pesquisadores declaram ainda que este trabalho foi realizado em concordância com o Código de Boas Práticas Científicas (FAPESP), não havendo nada em seu conteúdo que possa ser considerado como plágio, fabricação ou falsificação de dados. As opiniões, hipóteses e conclusões ou recomendações expressas neste material são de responsabilidade dos autores e não necessariamente refletem a visão da Fundação Pio XII – Hospital de Câncer de Barretos.” “Embora o Núcleo de Apoio ao Pesquisador do Hospital de Câncer de Barretos tenha realizado as análises estatísticas e orientado sua interpretação, a descrição da metodologia estatística, a apresentação dos resultados e suas conclusões são de inteira responsabilidade dos pesquisadores envolvidos. Os pesquisadores declaram não ter qualquer conflito de interesse relacionado a este estudo. DEDICATÓRIA A Deus, pelo dom da vida, de amar e me fortalecer a cada dia. Aos meus pais, Sandra M. Maciel Uzan e José Roberto Uzan por serem a minha referência como ser humano e como profissional e pelo incentivo insaciável para a minha formação. Aos meus irmãos Valéria e Márcio e aos meus sobrinhos Giovanna e Arthur pelo amor e apoio na realização dos meus sonhos. Ao meu namorado Alberto, pela força nas horas mais difíceis, pelo amor sincero, companheirismo e por participar ativamente da concretização deste trabalho com seu conhecimento e acreditando em mim. Dedico à vocês este trabalho. AGRADECIMENTOS Agradeço em primeiro lugar a Deus, pela saúde, força, e por estar comigo todos os dias, em todos os momentos incondicionalmente. Ao meu orientador, Prof. Dr. Luiz Fernando Lopes, por acreditar em mim como pessoa e como profissional, pelas palavras de incentivo durante as “tempestades” que encontramos neste caminho chamado mestrado e por contribuir para o meu crescimento profissional e intelectual. Ao meu co-orientador, Prof. Dr. Daniel Onofre Vidal, por acrescentar palavras de sabedoria nas linhas deste trabalho, pela exigência no meu aprendizado e na qualidade deste trabalho. Aos assessores das Bancas de Acompanhamento, Dr. Rui Reis e Dra. Ana Lucia Ambujamra que a cada encontro acrescentavam seus conhecimentos e sugestões para o aprimoramento deste trabalho. Ao Dr. Carlos Alberto Scrideli, pela pronta colaboração com este projeto. A Dra. Érica Boldrini, por me acompanhar na revisão dos prontuários e dividir seus conhecimentos comigo. A toda equipe do Núcleo de Apoio ao Pesquisador pelo suporte prestado, especialmente aos estatísticos Cleyton, Marco e Anderson por todo o aprendizado, e pela realização das análises metodológicas e estatísticas. Aos professores, colegas e equipe de suporte da Pós Graduação do Hospital de Câncer de Barretos que contribuíram para o meu aprendizado. Ao Dr. Valter Penna, pela coleta de todas as amostras de osteossarcoma que utilizamos neste projeto. Aos funcionários do Biobanco, em especial ao Dr. Cristovam Scapulatempo por revisar criteriosamente e pacientemente todas as lâminas do nosso estudo, e à minha amiga e excelente profissional Jacqueline, por me auxiliar em todas as etapas no laboratório. Ao Dr. Carlos Eduardo Bezerra, por avaliar os exames de imagem e redimensionar pacientemente os tumores avaliados. Ao André Van Helvoort Lengert, além do profissionalismo, agradeço imensamente pela amizade, pelas palavras de força e por contribuir ativamente da conclusão deste trabalho. Exemplo de humildade, simplicidade e inteligência. A Direção da Fundação Pio XII, em especial ao Sr. Henrique Duarte Prata, pelo incentivo aos estudos e permitindo que possamos nos ausentar em alguns (ou vários) momentos para a realização desse sonho. A minha Equipe de Farmacêuticos (34) e Auxiliares (63) da Farmácia da Fundação Pio XII – Hospital de Câncer de Barretos, pela compreensão nos momentos em que precisei me ausentar e pelas palavras de força nos momentos em que tudo parecia difícil. A minha família por ter lutado e muitas vezes se sacrificado para me dar esse privilégio de poder estar formada e realizar o sonho da Pós Graduação. Em especial, agradeço aos meus pais José Roberto Uzan e Sandra M. Maciel Uzan, por me ensinarem o amor ao próximo, a buscar os sonhos, a não desistir diante das dificuldades e pelo amor incondicional. Com muito amor e gratidão dedico esse trabalho a vocês. Ao meu médico particular, noivo e eterno namorado, Alberto Paiva de Moraes Filho, que durante esse período tornou-se mestre no assunto junto comigo e por acrescentar seus conhecimentos técnicos e científicos nesse trabalho. Meu companheiro de leituras de prontuários, interpretação de laudos de tomografias, raio-x e ressonâncias. Agradeço também por ser tão compreensivo, paciente, presente e incentivador. Obrigada pelas palavras de força e de ânimo nas horas certas! E por fim, aos pacientes, por nos conceder o uso de suas amostras para a realização deste trabalho. O sorriso que vemos no rosto de vocês diariamente, apesar das lutas que vocês enfrentam, é o que nos dá forças para continuar e não desistir. “ Seja lá o que for que você faça, empregue toda tua energia e todo teu espírito nesta tarefa. Ninguém conquista um sonho sem persegui-lo, ninguém anda uma milha sem dar o primeiro passo. Se ao fim da estrada alguma sombra de arrependimento te atacar, ainda assim, levante a cabeça, orgulhe-se por ter tentado, por ter buscado, por ter empregado todas as tuas forças até o último instante. Tanto pior e sempre pior é arrepender-se daquilo que você não fez” (Augusto Branco) “Eu tudo posso, naquele que me fortalece” Filipenses 4:13 SUMÁRIO 1 INTRODUÇÃO 1.1 Osteossarcoma 1 1.2 Alterações genéticas associadas ao osteossarcoma 3 1.3 Os RNAs não codificadores longos (lncRNAs) 5 1.4 Os lncRNAs associados aos processos de invasão e metástase em tumores humanos 8 1.4.1 MALAT1 (Metastasis associated lung adenocarcinoma transcript 1) 8 1.4.2 HOTAIR (HOX transcript antisense RNA) 9 1.4.3 HULC (Hepatocellular carcinoma up-regulated long non-coding RNA) 10 1.4.4 BCYRN1 (Brain cytoplasmic RNA1) 11 2 JUSTIFICATIVA 13 3 OBJETIVOS 3.1 Objetivo Geral 14 3.2 Objetivos Específicos 14 4 MATERIAIS E MÉTODOS 4.1 Amostras e seleção dos pacientes 15 4.2 Critérios de inclusão 15 4.3 Critérios de exclusão 15 4.4 Aspectos éticos 16 4.5 Coleta de dados 16 4.6 Avaliação da representatividade tumoral 16 4.7 Extração de RNA 17 4.8 Avaliação da quantidade e da qualidade do RNA total 18 4.9 Análise de contaminação do RNA com DNA genômico 19 4.10 Tratamento com DNase 19 4.11 Síntese de cDNA 20 4.12 Análise de expressão dos RNAs não codificadores longos por RT-qPCR 20 4.13 Análise estatística 5 RESULTADOS 5.1 Tamanho da amostra 23 5.2 Avaliação da representatividade tumoral 25 5.3 Extração, quantificação e avaliação da qualidade do RNA total 25 5.4 Avaliação de contaminação com DNA genômico e tratamento das amostras de RNA com DNAse 5.5 Análise da expressão dos normalizadores e requantificação das amostras utilizando o Qubit 5.6 21 27 28 Características clínicas, patológicas e demográficas dos pacientes com osteossarcoma 30 5.7 Análise da expressão dos lncRNAs 31 5.8 Associação da expressão dos lncRNAs com os fatores clínicopatológicos dos pacientes 5.9 33 Associação do perfil de expressão dos lncRNAs com as características clínico-patológicas dos pacientes 40 5.10 Análise de sobrevida global e sobrevida livre de eventos 47 6 DISCUSSÃO 53 6.1 Otimização do protocolo de extração e análise da qualidade do RNA 53 6.2 Caracterização das amostras 55 6.3 Expressão dos lncRNAs em osteossarcoma 58 6.3.1 Correlação da expressão do BCYRN1 com as características clínicopatológicas e sobrevida 6.3.2 Correlação da expressão do HOTAIR com as características clínicopatológicas e sobrevida 6.3.3 Correlação da expressão do HULC com as características clínicopatológicas e sobrevida 6.3.4 59 Correlação da expressão do MALAT1 com as características clínicopatológicas e sobrevida 60 62 63 6.4 Limitações do estudo 65 7 CONCLUSÕES 66 REFERÊNCIAS BIBLIOGRÁFICAS 66 ANEXOS Anexo A – Ficha de coleta de dados 78 Anexo B – Carta de aprovação do Comitê de Ética em Pesquisa do Hospital de Câncer de Barretos e do Hospital das Clínicas da Faculdade de Medicina de Ribeirão Preto/Universidade de São Paulo 81 LISTA DE FIGURAS Figura 1 - Funções celulares atribuídas aos diversos lncRNAs conhecidos 6 Figura 2 - Organização genômica dos lncRNAs 7 Figura 3 - Representação esquemática da estratégia desenvolvida neste trabalho para a avaliação da expressão de lncRNAs em osteossarcoma Figura 4 - Representatividade tumoral nas amostras selecionadas para a avaliação da expressão dos lncRNAs Figura 5 - 27 Expressão normalizada dos lncRNAs em osteossarcoma avaliada por RT-qPCR Figura 9 - 27 Gel de agarose para a avaliação da amplificação do gene KRAS nas amostras de RNA após tratamento com DNase Figura 8 - 26 Gel de agarose para a avaliação da amplificação do gene KRAS nas amostras de RNA Figura 7 - 25 Comparação da integridade do RNA de amostras de osteossarcoma após extração com dois protocolos diferentes Figura 6 - 24 32 Análise de sobrevida global e sobrevida livre de eventos para os pacientes metastáticos e não metastáticos ao diagnóstico tratados pelo protocolo GLATO 2006 no Departamento de Pediatria do Hospital de Câncer de Barretos no período de 2006 a 2013. Figura 10 - 47 Análise de sobrevida global de acordo com o perfil de expressão dos lncRNAs 51 Figura 11 - Análise de sobrevida livre de eventos de acordo com o perfil de expressão dos lncRNAs 52 LISTA DE TABELAS Tabela 1 - Condições de ciclagem para a PCR do gene KRAS Tabela 2 - Média e desvio-padrão dos Cts dos normalizadores ACTB e GAPDH, 19 obtidos após análise de expressão gênica a partir de RNA quantificado por meio do Nanovue Tabela 3 - Comparação da quantificação de RNA realizado pelos dois métodos utilizados neste trabalho Tabela 4 - 28 29 Média e desvio padrão dos Cts dos normalizadores ACTB e GAPDH, obtidos após análise de expressão gênica a partir de RNA quantificado por meio do Qubit Tabela 5 - Características clínicas, patológicas e demográficas dos pacientes com ostessarcoma para a análise de expressão dos lncRNAs Tabela 6 - 38 Comparação da média de expressão do MALAT1 com as características clínico-patológicas dos pacientes avaliados Tabela 10 - 37 Comparação da média de expressão do HULC com as características clínico-patológicas dos pacientes avaliados Tabela 9 - 36 Comparação da média de expressão do HOTAIR com as características clínico-patológicas dos pacientes avaliados Tabela 8 - 31 Comparação da média de expressão do BCYRN1 com as características clínico-patológicas dos pacientes avaliados Tabela 7 - 29 39 Área sob a curva, desvio padrão, p-valor e intervalo de confiança determinados pela curva ROC. 40 Tabela 11 - Ponto de corte, sensibilidade, especificidade, valor preditivo positivo (VPP), valor preditivo negativo (VPN) e acurácia, de acordo com a curva ROC. Tabela 12 - Associação do perfil de expressão do BCYRN1 com as caracterísiticas epidemiológicas e clínico-patológicas Tabela 13 - 48 Sobrevida livre de eventos (SLE) em 1 ano, 3 anos e 5 anos em relação às características clínico-patológicas Tabela 20 48 Sobrevida global (SG) em 1 ano, 3 anos e 5 anos em relação às características clínico-patológicas Tabela 19 - 47 Sobrevida livre de eventos (SLE) em 1 ano, 3 anos e 5 anos em relação às características epidemiológicas Tabela 18 - 46 Sobrevida global (SG) em 1 ano, 3 anos e 5 anos em relação às características epidemiológicas Tabela 17 - 45 Associação do perfil de expressão do MALAT1 com as caracterísiticas epidemiológicas e clínico-patológicas Tabela 16 - 44 Associação do perfil de expressão do HULC com as caracterísiticas epidemiológicas e clínico-patológicas Tabela 15 - 43 Associação do perfil de expressão do HOTAIR com as caracterísiticas epidemiológicas e clínico-patológicas Tabela 14 - 41 49 Sobrevida global (SG) em 1 ano, 3 anos e 5 anos em relação à expressão dos lncRNAs 49 Tabela 21 Sobrevida livre de eventos (SLE) em 1 ano, 3 anos e 5 anos em relação à expressão dos lncRNAs 50 LISTA DE ABREVIATURAS ACTB Actin beta ARF ADP-ribosylation factor ASXL1 Additional sex combs like 1 ASXL2 Additional sex combs like 2 ATRX Alpha thalassemia/mental retardation syndrome X-linked AUC Área sob a curva BAX BCL2-associated X protein BCYRN1 Brain cytoplasmic RNA 1 CASKI Linhagem celular de carcinoma epidermóide de cervix, HPV 16 CDKN2A Cyclin-dependent kinase inhibitor 2A CDKN2B Cyclin-dependent kinase inhibitor 2B cDNA DNA complementar CEP Comitê de Ética em Pesquisa CREB cAMP-response element-binding protein CREBBP cAMP responsive element binding protein Ct Cycle threshold CXCR4 C-X-C chemokine receptor type 4 DNA Ácido desoxiribonucléico DNase Desoxirribonuclease dNTP Desoxirribonucleotídeos fosfatados EUA Estados Unidos da América EZH2 Enhancer of zeste homolog 2 FGFR2 Fibroblast growth factor receptor 2 FMRP/USP Faculdade de Medicina de Ribeirão Preto/Universidade de São Paulo GAPDH Glyceraldehyde-3-phosphate dehydrogenase GBTO Grupo Brasileiro de Osteossarcoma H3F3A H3 histone, family 3ª HCB Hospital de Câncer Infantojuvenil de Barretos HIC1 Hypermethylated in cancer 1 HOTAIR HOX transcript antisense RNA HOXD Homeobox D HULC Hepatocellular carcinoma up-regulated long non-coding RNA IC Intervalo de confiança Kb Quilo bases KDM6A Lysine (K)-specific demethylase 6A KRAS Kirsten rat sarcoma viral oncogene homolog lncRNAs RNAs não codificadores longos MALAT1 Metastasis associated lung adenocarcinoma transcript 1 MAP2K4 Mitogen-activated protein kinase 4 MAPK7 Mitogen-activated protein kinase 7 MDM2 MDM2 proto-oncogene, E3 ubiquitin protein ligase MgCl2 Cloreto de magnésio miRNA Micro ácido ribonucleico MLL2 Myeloid/lymphoid or mixed-lineage leukemia 2 MMP9 Matrix metallopeptidase 9 mRNA Ácido ribonucléico mensageiro MYC Myelocytomatosis oncogene ncRNAs RNAs não codificadores NSD2 Nuclear SET domain-containing protein 2 OMS Organização Mundial de Saúde OS Osteossarcoma OSX Osterix PB Pares de bases PHF6 PHD finger protein 6 Phosphatidylinositol-4,5-biphosphate 3-kinase/thymoma viral PI3K/AKT/MTOR proto-oncogene/mechanism target of rapamycin (serine/threonine kinase) PRC2 Complexo repressivo polycomb 2 PRKACB cAMP-dependent protein kinase catalytic subunit beta RANKL Receptor activator of NF-kappa-B ligand RB1 Retinoblastoma RIN RNA Integrity number RNA Ácido ribonucléico RNA POL II RNA polimerase II RNase Ribonuclease ROC Receiver Operating Characteristics RT-qPCR Reação em cadeia da polimerase quantitativa SAME Serviço de Arquivo Médico Especializado SAOS2 Linhagem celular de osteossarcoma SETD2 SET domain containing 2 SG Sobrevida Global SisOnco Sistema Hospitalar Interno do Hospital de Câncer de Barretos SLE Sobrevida Livre de Eventos SMARCA4 SWI/SNF related, matrix associated, actin dependent regulator of chromatin, subfamily a, member 4 SMC1A Structural maintenance of chromosomes 1A SPSS 21 Statistical Package for Social Science TC Tomografia computadorizada TCLE Termo de Consentimento Livre e Esclarecido TP53 Tumor protein p53 TWIST Twist transcriptor factor USP7 Ubiquitin specific peptidase 7 ZMYM3 zinc finger, myocyte maintenance-type 3 LISTA DE SÍMBOLOS % Porcentagem ~ Aproximadamente < Menor ou igual a > Maior que < Menor que µm Micrômetro mg Miligrama mL Mililitro rpm Rotações por minuto µL Microlitro G Gravidade (unidade de rotação em centrífuga) °C Graus Celsius nm Nanômetro ng Nanograma mM Milimolar µM Micromolar U Unidades µg Micrograma cm Centímetros > Maior ou igual a P Valor de p vs versus RESUMO Uzan VRM. A Expressão gênica dos RNAs não codificadores HOTAIR, MALAT1, HULC E BCYRN1 em osteossarcoma. Dissertação (Mestrado). Barretos: Hospital de Câncer de Barretos; 2015. JUSTIFICATIVA: Os tumores ósseos primários representam 5-6% de todos os tumores da infância e adolescência (0-19 anos), sendo o osteossarcoma o câncer ósseo primário mais comum nesta faixa etária. Cerca de 20% dos pacientes apresentam metástases ao diagnóstico e, em torno de 40% evoluirão com metástase, resultando em uma baixa taxa de sobrevida de 20% em cinco anos. Geneticamente, o osteossarcoma é um tumor extremamente complexo e por isso, torna-se necessária a busca de marcadores moleculares que possam indicar o prognóstico, ou mesmo a presença de metástases ao diagnóstico visando o auxílio do tratamento desses pacientes. OBJETIVO: Os objetivos deste estudo foram otimizar um protocolo de extração e avaliação da qualidade do RNA em osteossarcoma, avaliar a expressão gênica de quatro RNAs não codificadores longos (HOTAIR, MALAT1, HULC E BCYRN1) em amostras de osteossarcoma primário e avaliar o potencial dessas moléculas como marcadores de prognóstico de pacientes com osteossarcoma. MATERIAIS E MÉTODOS: Inicialmente, 175 pacientes portadores de osteossarcoma foram identificados no banco de dados da nossa instituição. Após rigorosos critérios de inclusão e exclusão, 36 amostras pré-quimioterapia, provenientes de pacientes diagnosticados entre 2006 e 2013, foram incluídas no nosso estudo. Todas as amostras de osteossarcoma foram revisadas por um patologista e àquelas que apresentaram pelo menos 50% de tecido tumoral foram submetidas a extração do RNA. Após a extração, as amostras foram analisadas quanto a integridade do RNA, quantificadas e a expressão dos genes foi avaliada por meio da RT-PCR em tempo real. RESULTADOS: No nosso trabalho a média de idade dos pacientes foi de 15 anos, sendo a maioria (55,6%) do sexo feminino. A localização mais frequente do tumor foi o fêmur (44,4%) e o subtipo histológico osteoblástico foi predominante (77,8%). A maioria dos pacientes no estudo apresentou resposta parcial à quimioterapia (72,2%) e 44,9% dos pacientes apresentaram metástases ao diagnóstico ou durante o tratamento. Em relação a expressão dos lncRNAs, nossos resultados mostraram que existe uma ampla variação na expressão dos quatro genes propostos entre as amostras e que a média de expressão maior dos genes BCYRN1 e MALAT1 foi associada significativamente a idade superior a 10 anos. Após categorização da expressão, observamos que a maioria dos pacientes que apresentaram baixa expressão do gene BCYRN1 pertenciam ao grupo do tipo histológico osteoblástico. Para o gene HOTAIR, a alta expressão foi associada à pacientes com tumor menor que 12 cm ao diagnóstico, e para o MALAT1, a alta expressão foi observada em pacientes com idade ao diagnóstico igual ou superior a 10 anos. CONCLUSÕES: Nossos achados demostraram que a maior expressão de BCYRN1 e MALAT1 foi associada significativamente a pacientes com idade acima de 10 anos. Não encontramos associação significativa entre a expressão dos genes e a sobrevida dos pacientes. Nossos dados em conjunto sugerem que a expressão de BCYRN1 e HOTAIR merecem uma investigação mais abrangente em OS. PALAVRAS-CHAVE: Osteossarcoma; RNAs não codificadores longos; Metástase; Expressão Gênica; PCR em Tempo Real; Biomarcadores. ABSTRACT Uzan VRM. The gene expression of non-coding RNAs HOTAIR, MALAT1, HULC and BCYRN1 in osteosarcoma. Dissertation (Master’s degree). Barretos: Barretos Cancer Hospital, 2015. BACKGROUND: The primary bone tumors represent 5-6% of all tumors of childhood and adolescence (0-19 years), and osteosarcoma is the most common primary bone cancer in this age group. About 20% of patients present metastases at diagnosis, and approximately 40% will develop metastatic disease, resulting in low survival rate of 20% in five years. Genetically, the osteosarcoma is an extremely complex tumor, therefore it becomes necessary to identify new molecular markers that may predict the prognosis or the presence of metastasis at diagnosis in osteosarcoma patients. OBJECTIVES: Standardize a protocol for RNA extraction and quality assessment, evaluate the expression of non-coding RNAs (MALAT1, HOTAIR, HULC and BCYRNA1) in primary osteosarcoma samples and also to evaluate the potential role of these molecules as prognostic markers in osteosarcoma. MATERIAL AND METHODS: Initially, 175 patients with osteosarcoma were identified in the database of our institution. After stringent criteria for inclusion and exclusion, 36 prechemotherapy samples, from patients diagnosed between 2006 and 2013 were included in our study. The RNA extraction was performed using the TRIZOL method and, in some specific cases, using commercial kit. After extraction, the samples were analyzed for their integrity, quantified, and the gene expression was assessed by real time RT-PCR. RESULTS: The average age of the patients was 15 years, being the majority (55,6%) female. The most frequent tumor site was femur (44,4%) and the osteoblastic histologic subtype was predominant (77,8%). The majority of the patients presented partial response to treatment (72,2%) and almost half (44.9%) presented metastasis at diagnosis or during treatment. Regarding the expression of lncRNAs, our results showed that there is a wide variation in the expression of the proposed four genes between samples and the average higher expression of BCYRN1 and MALAT1 genes was significantly associated with the age of 10 years. After categorization of expression in high and low, we observed that most patients with low expression of the gene BCYRN1 belonged to the osteoblastic histological type group. For HOTAIR gene overexpression has been associated with patients with lower than 12 cm tumor diagnosis, and for MALAT1, high expression was observed in patients with age at diagnosis less than 10 years. CONCLUSIONS: Our findings showed that the highest expression of BCYRN1 and MALAT1 was significantly associated with patients over the age of 10 years. We found no significant association between gene expression and patient survival. Our data together suggested that the expression of BCYRN1 and HOTAIR deserves a comprehensive analysis in OS. KEYWORDS: Osteosarcoma; long non-coding RNAs; Metastasis; Gene Expression; Real-Time Polymerase Chain Reaction; Biomarker. 1 1 INTRODUÇÃO 1.1 Osteossarcoma Os tumores ósseos primários representam 5-6% de todos os tumores da infância e adolescência (0-19 anos), sendo o osteossarcoma (OS) o câncer ósseo primário mais comum nesta faixa etária. O OS apresenta uma distribuição bimodal de idade, sendo o primeiro pico observado na segunda década de vida (14-19 anos) e o segundo pico a partir da sexta década de vida (60 anos)1. Em geral, a incidência na população é de 2-4 casos/milhão ao ano, entretanto na adolescência (15-19 anos) a incidência pode chegar a 8-10 casos/milhão ao ano. O OS representa de 10 a 15% dos tumores sólidos extracraniais nesta faixa etária 1, 2. Nos Estados Unidos (EUA) são esperados aproximadamente 400 novos casos de OS por ano, em indivíduos até 20 anos3. No Brasil, a maior incidência deste tipo de tumor está registrada no estado de São Paulo4, no qual são descritos 648 casos (~ 54 casos/ano) de OS entre 2000 e junho de 2012, em pacientes de 0 a 18 anos5. No Hospital de Câncer Infantojuvenil de Barretos (HCB) há registro de 225 casos de OS entre os anos 2000 e 2013. O OS é um tumor maligno definido pela presença de células mesenquimais alteradas que produzem osteóide e/ou tecido ósseo imaturo6. Se origina mais frequentemente na região metafisária dos ossos longos, no interior da cavidade medular e penetra no córtex ósseo, envolvendo e invadindo os tecidos moles adjacentes7. Na classificação atual da Organização Mundial de Saúde (OMS), o OS pode ser dividido em: convencional, telangectásico, de pequenas células, central de baixo grau, secundário, parosteal, periosteal e, por último, de superfície de alto grau. O diagnóstico histológico do tipo convencional compreende a grande maioria deste tipo de tumor e é dividido em três subtipos: osteoblástico, condroblástico e fibroblástico, dependendo do tipo predominante de matriz presente no tumor6. O subtipo histológico não tem impacto sobre a resposta à quimioterapia e os pacientes recebem o mesmo tratamento, independentemente do subtipo histológico, sugerindo que estes vários padrões de diferenciação são reflexo de uma única entidade clínica8. Historicamente, o prognóstico de pacientes com OS tratados apenas com cirurgia e/ou radioterapia era ruim, com sobrevida em torno de 20% em dois anos9. A maioria desses pacientes iria ao óbito devido à evolução da doença e estabelecimento de metástases 2 pulmonares6. A introdução da quimioterapia sistêmica no tratamento do OS resultou em uma melhora significativa no prognóstico dos pacientes, elevando a sobrevida livre de eventos a 60 – 70%, em três anos, nos casos de OS localizados de extremidades10-12. Aproximadamente, 15-20% dos pacientes com OS apresentam metástases ao diagnóstico, detectadas por exames radiológicos, como a tomografia computadorizada (TC) e, adicionalmente em torno de 40% evoluirão com metástase em fases posteriores durante o tratamento12, 13. As metástases são muito frequentes nos pulmões (~80%), mas também podem ocorrer em outros sítios ósseos, medula óssea e, raramente, em linfonodos. Os sítios metastáticos da doença comumente apresentam resistência ao tratamento, resultando em uma baixa taxa de sobrevida do paciente, em torno de 20% em cinco anos12, 14, 15. O tratamento para o osteossarcoma preconiza a utilização combinada de quimioterapia em altas doses, ressecção da lesão primária e de todos os sítios metastáticos da doença, geralmente por toracotomia exploratória com apalpação de todo pulmão 16. Na busca de novas alternativas para o sucesso no tratamento do osteossarcoma, o Grupo Brasileiro para Tratamento de Osteossarcoma (GBTO)17, posteriormente Grupo Latino americano de Oncologia Pediátrica (GALOP), vem publicando estudos propondo modificações nos esquemas de tratamento quimioterápico a fim de melhorar a sobrevida desses pacientes. Entretanto, os resultados encontrados mostraram que mesmo intensificando o tratamento não houve melhora no grau de necrose ou na taxa de sobrevivência17. Aproximadamente, 30% de todos os pacientes com metástase pulmonar ao diagnóstico e, mais do que 40% daqueles que alcançarem uma remissão cirúrgica completa podem apresentar bom prognóstico a longo prazo15. O tratamento para recidiva local ou nos pulmões é inicialmente cirúrgico. A recorrência de metástase pulmonar é frequente e, embora o prognóstico destes pacientes seja ruim, com uma sobrevida pós-recaída de 20% em cinco anos, a retirada da lesão deve ser realizada quando viável18. Em contraste, quando isso é possível em torno de 33% dos pacientes com múltiplas recorrências ressecáveis sobrevivem cincos anos ou mais19. Assim, a detecção da metástase ao diagnóstico ou mesmo o acompanhamento da lesão metastática é essencial no tratamento do OS. Atualmente, a técnica padrão utilizada na detecção e seguimento da metástase pulmonar é TC de tórax. Entretanto, tal técnica tende a subestimar o número de metástases pulmonares e também pode falhar na 3 identificação de metástases ao diagnóstico, o que pode levar a um tratamento inadequado destes pacientes19. 1.2 Alterações genéticas associadas ao osteossarcoma Geneticamente, o OS é um tumor extremamente complexo, não havendo uma alteração específica identificada neste tipo de sarcoma. Apesar dessa complexidade genética, existem numerosos achados quanto às alterações genéticas que podem estar associadas à patogênese do OS20. A descoberta de tais alterações foi originada de estudos que avaliaram modelos animais (rato) de OS, síndromes de predisposição genética, fatores etiológicoambientais e a abordagem direta destes tipos de tumores21. Diversos modelos animais que apresentam diferentes alterações genéticas desenvolvem OS. Como exemplo, podemos citar os animais com alterações em Tp53, dos quais cerca de 40-50% desenvolvem OS22, 23; em Bax e Arf, dos quais 44%24; em Hic1, dos quais 40%25; entre outros. Entre as síndromes familiais que predispõe os indivíduos ao desenvolvimento de OS pode-se destacar a síndrome de Li-Fraumeni e do Retinoblastoma, apresentando alterações nos genes Tp53 e Rb1, respectivamente26, 27. Em relação aos fatores ambientais, agentes físicos, químicos e biológicos têm sido sugeridos como carcinógenos para o OS. Entre eles, a ação da radiação ionizante já tem sido associada ao maior risco de desenvolvimento de OS28. Uma variedade de genes também é descrita com alterações nos OS que ocorrem de forma esporádica. Entre eles podemos citar: TWIST, MYC, CDKN2A, CDKN2B, FGFR2, MDM2, MAPK7, MAP2K4, MMP9, entre muitos outros29. TesserGamba et al.30 estudaram a expressão de 10 genes localizados em regiões de rearranjo cromossômico em OS. Dentre eles, encontravam-se os genes MAPK7 e MAP2K4 que estavam altamente expressos na maioria das amostras analisadas e a expressão desses genes foi significativamente associada com a presença de metástase ao diagnóstico, baixa resposta à quimioterapia e pior sobrevida global. Com o intuito de encontrar alvos moleculares para as terapias existentes ou em desenvolvimento, um estudo realizado com pacientes após recaída de OS avançado, encontrou comumente alterações na via PI3K/Akt/mTOR e sugeriu que existe uma elevada diversidade molecular nesta doença e a necessidade de mais estudos para definir subgrupos distintos de alterações genéticas31. 4 Um estudo que avaliou alterações em 633 genes reguladores epigenéticos em amostras de 1000 tumores pediátricos diversos revelou que 72 genes estavam recorrentemente mutados em todo o grupo de pacientes estudados. Os pacientes portadores de OS apresentaram altas taxas de mutação nos genes reguladores epigenéticos, tais como, H3F3A, PHF6, ATRX, KDM6A, SMARCA4, ASXL2, CREBBP, EZH2, MLL2, USP7, ASXL1, NSD2, SETD2, SMC1A e ZMYM332. Outro estudo teve como objetivo mostrar o comportamento metastático do OS através da expressão dos genes e da análise citogenética e encontrou que os genes MDM2, CXCR4, RANKL, RB1 e OSX possuem expressões modificadas de acordo com o momento do surgimento da metástase (ao diagnóstico ou durante o tratamento), podendo haver perda do RANKL ou ganho do CXCR4 em tumores de pacientes metastáticos ao diagnóstico. Eventos como esses, facilitam a progressão da metástase concomitante com o estabelecimento do tumor primário e apoiam o papel do receptor CXCR4 em direcionar a metástase para o pulmão. Por outro lado, acontecimentos tardios, como a perda do RB1 e ganho de MDM2, que são genes reguladores fundamentais do ciclo celular, parece estar relacionada com os mecanismos finais que contribuem para o estabelecimento da metástase no OS33. Alterações cromossômicas também são frequentes no OS. A utilização de técnicas convencionais de citogenética tem demonstrado como as mais comuns o ganho do cromossomo 1 e as perdas dos cromossomos 2, 6, 8, 9, 10, 13 e 17 34, 35. Ainda, rearranjos cromossômicos foram descritos envolvendo os cromossomos 1, 4, 6, 7, 11, 12, 14, 15, 17, 19 e 2236, 37. Esses dados sugerem que a instabilidade genômica é frequente e importante para o desenvolvimento do OS e envolve vias que normalmente mantém a integridade genômica. Apesar de todo esse conhecimento sobre as alterações moleculares envolvidas na patogênese do OS, a doença metastática ainda é um desafio, sendo o processo metastático pouco compreendido na evolução do OS. Dessa forma, há uma necessidade crítica de identificar marcadores que possam indicar com precisão ou predizer a existência de doença metastática em pacientes com OS, uma vez que a maioria desses pacientes apresenta doença micrometastática não detectável pelos exames de imagem ao diagnóstico. Além disso, estratificar os pacientes de maneira correta pode auxiliar no correto manejo do tratamento, principalmente nos pacientes não metastáticos, que podem ser beneficiados com tratamentos menos tóxicos sem piora na resposta ou prognóstico. 5 1.3 Os RNAs não codificadores longos (lncRNAs) Nos últimos anos, principalmente com o desenvolvimento de novas técnicas como o sequenciamento em larga escala e os DNA tiling arrays, houve uma revolução na nossa visão em relação à organização e conteúdo do genoma humano. Previamente ao que era assumido, tais técnicas revelaram o achado inesperado de que a maior parte do genoma humano é transcrito em RNA. Atualmente, estima-se que mais de 70% do genoma humano seja transcrito, entretanto apenas 2% servem de modelo para a produção de proteínas 38, 39. Assim, as moléculas de RNA que não possuem a capacidade de codificar proteínas são denominadas como RNAs não codificadores (ncRNAs). De acordo com o seu tamanho, os ncRNAs são divididos em duas classes principais: os ncRNAs pequenos (< 200 nucleotídeos) e os ncRNAs longos (lncRNAs > 200 nucleotídeos). Recentemente, os ncRNAs pequenos têm recebido mais atenção, especialmente os microRNAs (miRNAs) que têm sido descritos com um papel crucial no desenvolvimento de diversas doenças, incluindo o câncer40. Entretanto, torna-se cada vez mais claro o fato de que o genoma de mamíferos também codifica vários lncRNAs, definidos como RNAs endógenos com tamanho superior a 200 nucleotídeos que não apresentam uma significativa fase aberta de leitura (menos de 100 aminoácidos), dessa forma não resultando em proteína41. A maioria dos lncRNAs descritos até o momento é transcrito pela RNA polimerase II (RNA pol II) e são poliadenilados42, entretanto em alguns casos, como o BCYRN1, apesar de funcionais os lncRNAs não são poliadenilados43. Assim, os lncRNAs constituem um grupo heterogêneo de moléculas de RNA e podem atuar de diversas maneiras diferentes em funções biológicas e moleculares nas células. Dentre suas diversas funções, em geral os lncRNAs podem regular a expressão gênica44, influenciar a localização das proteínas45 e, ainda, são importantes para a formação de subestruturas celulares ou complexos proteicos essenciais para as células46. A figura 1 demonstra as funções celulares de alguns dos lncRNAs já descritos. 6 Fonte: Gutschner & Diederichs47 Figura 1. Funções celulares atribuídas aos diversos lncRNAs conhecidos. A) lncRNAs podem ser processados em ncRNAs pequenos que podem alvejar outros RNAs, o que subsequentemente leva a degradação do RNA alvo; B) lncRNAs podem atuar como “esponjas” de miRNAs, assim inibindo sua função e influenciando na expressão dos RNAs alvos do miRNA; C e H) lncRNAs são importantes na formação de estruturas subcelulares e complexos de proteínas, nos quais desempenham uma função de arcabouço; D) lncRNAs podem interagir com proteínas alterando sua função e localização celular; E) lncRNAs regulam a expressão gênica pelo recrutamento de fatores de transcrição a região promotora dos seus genes alvos; F) lncRNAs podem regular o mecanismo de splicing alternativo de pré-mRNAs; G) lncRNAs podem interagir com complexos de remodelamento de cromatina e induzir alterações locais ou globais no “empacotamento” da cromatina. Os lcnRNAs frequentemente se sobrepõem com ou estão intercalados a transcritos codificantes e não codificantes42. Em termos de posição genômica, os lncRNAs podem ser agrupados em cinco diferentes categorias: (1) senso ou (2) antisenso, quando sobrepõe um ou mais exons de outro transcrito na mesma fita ou na fita oposta de DNA, respectivamente; (3) bidirecional, quando sua expressão e a de um transcrito adjacente na fita oposta é iniciada em regiões genômicas próximas; (4) intrônico, quando se origina de um intron de um segundo transcrito e; (5) intergênico, quando ocorre como uma unidade independente dentro do intervalo genômico entre dois genes (Figura 2). 7 Fonte: criado por Vidal DO, 2013. Figura 2. Organização genômica dos lncRNAs. A linha preta representa a fita de DNA; As caixas pretas representam os exons, a linha preta pontilhada representa a posição dos introns e as setas indicam a direção de transcrição dos genes. Muitos estudos vêm sendo desenvolvidos no intuito de identificar novos lncRNAs. Um dos relatos mais recentes identificou 5.440 lncRNAs no genoma humano que quando combinados com o resultado de outros quatro estudos resultou em aproximadamente 7.000 lncRNAs48. Estimativas recentes levam a crer que existam mais de 20.000 lncRNAs transcritos no genoma humano, implicando que esta classe de lncRNAs representará um enorme, ainda desconhecido, componente de redes celulares normais que pode estar alterado na biologia da célula tumoral49. 8 De fato, os lncRNAs são frequentemente expressos de forma específica seja em uma doença, tecido ou estágio de desenvolvimento, o que torna tais moléculas alvos atrativos a novas terapias voltadas às funções específicas dos lncRNAs tanto no desenvolvimento quanto em doenças50, 51. No entanto, nossos conhecimentos de como os lncRNAs podem agir nas células e quais funções eles podem ter nas doenças, como o câncer, ainda é muito limitado. 1.4 Os lncRNAs associados aos processos de invasão e metástase em tumores humanos Uma das principais características do câncer é a sua habilidade de invadir tecidos adjacentes e promover a formação de metástases à distância, é também um dos maiores desafios no tratamento oncológico e implica na interação e regulação de complexos mecanismos biológicos. Frequentemente, as células tumorais passam por alterações morfológicas, hoje conhecidas como transição epitélio-mesênquima, modificando sua interação célula-célula ou célula-matriz. Portanto, toda essa cascata de invasão e metástase compreende múltiplas alterações biológicas que permitem as células invadirem tecidos sadios, seguido da intravasão nos vasos sanguíneos e linfáticos. Durante este trânsito na corrente sanguínea, as células escapam da vigilância do sistema imune e adquirem a capacidade de crescimento independente de ancoragem, além de extravasarem nos tecidos alvos e promover a instalação de micrometástases e posteriormente o crescimento de um tumor52. Neste contexto, alguns lncRNAs têm sido descritos como tendo um papel fundamental no processo metastático. Dentre eles podemos destacar o MALAT1, HOTAIR, HULC e BCYRN1. 1.4.1 MALAT1 (Metastasis associated lung adenocarcinoma transcript 1) Um dos primeiros lncRNAs a ser descrito com tal papel foi o MALAT1, um transcrito de 8kb, localizado no cromossomo 11q13.1 que é um ponto de quebra frequente de translocações cromossômicas associadas ao câncer53-55. MALAT1 fica retido no núcleo e está localizado nos speckles nucleares, que são estruturas envolvidas no processamento do prémRNA56. Recentemente, MALAT1 foi demonstrado atuando na regulação do splicing alternativo de pré-mRNAs pela modulação dos níveis de fatores de splicing. A supressão de MALAT1 altera o processamento de um conjunto de pré-mRNAs, os quais desempenham um 9 papel importante na biologia do câncer. Esse gene também é altamente conservado entre diversas espécies ressaltando sua importância funcional53. Primeiramente, o MALAT1 foi descrito com um aumento de expressão no tumor primário, como um marcador prognóstico de metástase e pior sobrevida de pacientes acometidos pelo carcinoma de pulmão de células não pequenas. Essa associação com a metástase se mostrou estágio e histologia específica, dessa forma servindo como fator prognóstico independente na sobrevida do paciente em estágios iniciais do adenocarcinoma de pulmão57. A expressão de MALAT1 foi observada em diversos tecidos normais, com os maiores níveis de expressão em pâncreas e pulmão57, entretanto sua expressão está aumentada em uma gama de outros tumores quando comparados aos tecidos normais correspondentes como, por exemplo, no carcinoma hepatocelular58. MALAT1 pode estar associado com parâmetros clínicos e pode controlar a proliferação celular, a apoptose, a migração, invasão, ou a disseminação metastática nas células tumorais59. Um estudo demonstrou que o MALAT1 promove a mobilidade celular em células tumorais de pulmão, por meio da regulação transcricional ou pós-transcricional de genes importantes para este mecanismo celular60. Adicionalmente, MALAT1 suporta a proliferação e invasão de células de câncer cervical (CaSki), e sua inibição leva ao aumento de expressão de proteínas pró-apoptóticas como caspase 3, 8 e Bax61. A expressão de MALAT1 também foi avaliada em pacientes com mieloma e mostrou estar superexpresso em pacientes recém diagnosticados em comparação com pacientes pós tratamento. A expressão desse gene foi relacionada com o status da doença e a mudança da expressão pós tratamento teve relevância prognóstica. Pacientes que sofreram progressão precoce tiveram uma alteração significativamente menor na expressão de MALAT1 após tratamento. Sendo assim, MALAT1 mostrou ter um papel fundamental na patogênese do mieloma62. Recentemente, MALAT1 foi associado com o processo de proliferação e metástase em osteossarcoma, através da ativação da via PI3K/Akt. Esses resultados indicaram que MALAT1 pode ser considerado como alvo terapêutico no osteossarcoma humano63. 1.4.2 HOTAIR (HOX transcript antisense RNA) O HOTAIR é um gene de 2.2 kb localizado no cromossomo 12q13.13. HOTAIR atua na interação e recrutamento do complexo repressivo polycomb 2 (PRC2) no locus do gene 10 HOXD o que leva ao silenciamento transcricional de 40 kb a montante44. Devido a sua importância na regulação epigenética da expressão gênica, não é surpreendente que a expressão de HOTAIR esteja alterada em uma série de tumores. Em um dos primeiros relatos, a expressão aumentada de HOTAIR foi associada a tumores primários de mama em relação ao tecido normal. Ainda, o seu nível de expressão nos tumores primários correlacionou-se positivamente com a ocorrência de metástases e pior prognóstico nestes pacientes. A superexpressão de HOTAIR em células epiteliais cancerosas levou ao aumento do potencial de invasão e formação de metástases destas células. Em contrapartida, a supressão de HOTAIR levou a inibição de tais processos biológicos64. Resultados semelhantes foram descritos em carcinoma hepatocelular, no qual a expressão de HOTAIR também está aumentada em tumores primários e parece ser um biomarcador potencial para ocorrência de metástases em linfonodos. Ainda, a expressão de HOTAIR mostrou-se um fator independente de prognóstico associado à recorrência da doença e sobrevida menor dos pacientes. A depleção de HOTAIR em células tumorais do fígado reduz a capacidade de invasão e viabilidade dessas células65. Somado a isso, a supressão de HOTAIR torna as células cancerígenas mais sensíveis à ação de quimioterápicos como a cisplatina e doxorrubicina66. A alta expressão do HOTAIR ainda está relacionada ao potencial invasivo e metastático do adenocarcinoma gástrico67, do melanoma68, do carcinoma endometrial69 e do câncer de colo de útero70, entre outros. Xue et al.71 identificaram variantes genéticas do HOTAIR que se associam a um risco aumentado de câncer colorretal. Meta-análises recentes confirmam a associação entre a expressão elevada de HOTAIR com prognóstico desfavorável e sobrevida reduzida em um grande número de malignidades72, 73. 1.4.3 HULC (Hepatocellular carcinoma up-regulated long non-coding RNA) O HULC é um gene de 500kb localizado no cromossomo 6p24.3. HULC foi inicialmente descrito como um ncRNA específico do fígado que é altamente expresso em tumores primários do fígado74. Também apresenta expressão elevada em metástases hepáticas de pacientes com carcinoma colorretal, entretanto não é expresso nestes tumores primários 75. Recente relato demonstrou que HULC funciona como uma “esponja” de miRNAs, se ligando e provocando a diminuição da expressão de uma série de miRNAs, incluindo o miR-372. A redução na expressão deste miRNA leva ao aumento da expressão do seu alvo PRKACB, que 11 por sua vez induz a fosforilação de CREB. Isso resulta na alteração dos processos de deacetilação e metilação de histonas, gerando um impacto no remodelamento da cromatina alterando assim a expressão de uma série de genes76. Um estudo comparou a expressão tecidual e plasmática do HULC entre pacientes com hepatocarcinoma e um grupo controle. Além de serem constatados níveis significativamente maiores de HULC no tecido/plasma de pacientes com hepatocarcinoma, houve relação direta entre os valores de expressão do HULC e outras variáveis, como o grau de diferenciação histológica. Estes resultados sugerem um potencial uso deste RNA como biomarcador diagnóstico e/ou prognóstico77. Um trabalho mostrou superexpressão do HULC em tecidos de câncer gástrico, notando-se associação direta com estádios mais avançados e presença de metástases. O mesmo trabalho estudou os efeitos da superexpressão e do “knockdown” da expressão do HULC em células, determinando que o HULC desempenha papel importante na proliferação, migração e invasão celular, bem como na inibição da apoptose78. Outro estudo investigou a expressão do HULC em tecidos de câncer de pâncreas, observando-se correlação significativa entre maior expressão do gene com tamanho tumoral, invasão vascular e o estádio da doença79. 1.4.4 BCYRN1 (Brain cytoplasmic RNA 1) O BCYRN1, também conhecido como BC200, é um gene de 200pb localizado no cromossomo 2p21, que é especificamente expresso em tecidos do sistema nervoso. Sua expressão já foi descrita em células germinativas, entretanto normalmente não é expresso em tecidos somáticos de origem não neural. Uma análise em 19 tipos diferentes de tumor revelou sua expressão nos carcinomas de mama, colo do útero, esôfago, pulmão e ovário, mas não nos tecidos normais correspondentes80. Em outro trabalho, os autores identificaram a expressão elevada de BCYRN1 em carcinoma invasivo de mama, mas não em tecido normal. Uma análise de sensibilidade e especificidade confirmou o poder prognóstico da expressão do BCYRN1 como um marcador molecular deste tipo de tumor. Além disso, em carcinoma ductal in situ, a expressão de BCYRN1 foi relacionada com alto grau nuclear, apontando que possa ser utilizado como marcador prognóstico de progressão tumoral 43. Tais exemplos deixam evidente o importante papel funcional dos lncRNAs para a ativação do mecanismo de invasão celular e formação de metástases. De fato, os lncRNAs 12 descritos podem regular uma gama de genes envolvidos nestes processos. Por fim, lncRNAs especificamente expressos ou silenciados em tumores humanos podem desenvolver um papel importante no estabelecimento do fenótipo tumoral e, portanto, representar um grupo de potenciais novos alvos terapêuticos no combate ao câncer. Visto isso, nós avaliamos a expressão dos lncRNAs MALAT1, HOTAIR, HULC e BCYRN1 em amostras primárias de osteossarcoma ao diagnóstico e o potencial papel destas moléculas como marcadores prognósticos nesta doença. 13 2 JUSTIFICATIVA Os lncRNAs estão associados a regulação da expressão gênica e podem atuar por diferentes mecanismos. Os lncRNAs MALAT1, HOTAIR, HULC e BCYRN1 já foram associados a diversos fatores prognóstico como presença de metástase, baixa resposta à quimioterapia e o processo metastático em tumores humanos. Nossa hipótese é de que esses genes também possam estar envolvidos com a progressão do tumor, resistência ao tratamento e o processo metastático em osteossarcoma. Como estes tumores são muito agressivos e metade dos pacientes apresenta metástase ao diagnóstico ou durante o tratamento, com um prognóstico muito pobre, a avaliação dessas moléculas pode indicar novos marcadores biológicos com potencial prognóstico para este tumor, bem como novos alvos para terapias alternativas no osteossarcoma. 14 3 3.1 OBJETIVOS Objetivo geral Avaliar a expressão gênica de MALAT1, HOTAIR, HULC e BCYRN1 em amostras de osteossarcoma e avaliar o potencial papel destes genes como marcadores prognósticos nesses pacientes. 3.2 Objetivos específicos Otimizar um protocolo de extração e análise de qualidade de RNA em osteossarcoma; Correlacionar a expressão dos genes com os dados clínico-patológicos dos pacientes; Analisar a sobrevida global e sobrevida livre de eventos dos pacientes de acordo com a expressão dos genes avaliados. 15 4 4.1 MATERIAIS E MÉTODOS Amostras e seleção dos pacientes O levantamento dos pacientes diagnosticados com OS entre os anos de 2006 e 2013 foi realizado por meio do SisOnco, um sistema informatizado, o qual contém a base de dados da patologia do Hospital de Câncer de Barretos (HCB). Nesse período, foram identificados 175 pacientes com OS. Ainda, por meio de uma colaboração estabelecida com o Dr. Carlos Alberto Scrideli, 32 amostras de RNAs de OS provenientes do Banco de Tumores do Laboratório de Pediatria da Faculdade de Medicina de Ribeirão Preto – Universidade de São Paulo (FMRP/USP) foram incluídas em nossas análises. Além das amostras de osteossarcoma, também avaliamos expressão dos genes de interesse em uma linhagem celular não tumorigênica de osteossarcoma (SAOS). Para a avaliação dos critérios de elegibilidade, foi utilizada uma ficha de coleta de dados (Anexo A), contendo 62 parâmetros. Através dos dados obtidos em prontuário, a ficha foi preenchida e criou-se um banco de dados no programa SPSS 21. 4.2 Critérios de inclusão Os participantes deste estudo atenderam aos seguintes critérios de inclusão: Qualquer idade ao diagnóstico; Diagnóstico definido de osteossarcoma (ósseo), de qualquer localização anatômica e histologia; Tratamento realizado pela equipe de Pediatria; Com metástase ou não ao diagnóstico; Amostras criopreservadas disponíveis para a extração de RNA. 4.3 Critérios de exclusão Os critérios de exclusão deste estudo foram: Pacientes cujo início do tratamento quimioterápico ocorreu anteriormente a coleta do material biológico; Amostras que não possuíam representatividade de tumor maior que 50%; Amostras cuja integridade do RNA estava comprometida (RIN < 4.0); Diagnóstico de osteossarcoma como 2º tumor primário. 16 4.4 Aspectos éticos O projeto foi encaminhado ao Comitê de Ética e Pesquisa (CEP) da Fundação Pio XII – HCB (aprovado em abril de 2013 – número de referência 251.134) e do Hospital das Clínicas da Faculdade de Medicina de Ribeirão Preto da Universidade de São Paulo (FMRP/USP) (aprovado em setembro de 2014), de modo a atender as exigências da Resolução nº 466/2012 do Conselho Nacional de Saúde/Ministério da Saúde (Anexo B). A dispensa do Termo de Consentimento Livre e Esclarecido (TCLE) foi concedida pelo CEP por se tratar de um estudo com uso de amostras retrospectivas e de risco mínimo para os participantes da pesquisa, sendo este risco inerente a quebra acidental do sigilo de informações. Os pesquisadores envolvidos se comprometeram a garantir o sigilo de todos os pacientes, não divulgando publicamente o nome ou qualquer outra informação que possa identificar os pacientes envolvidos neste estudo. 4.5 Coleta dos dados A coleta dos dados clínicos-patológicos foi realizada no SAME (Serviço de Arquivo Médico Especializado) na Fundação Pio XII – HCB e no mesmo departamento da Fundação Pio XII - Hospital de Câncer Infantojuvenil de Barretos. Os dados clínico-patológicos das amostras provenientes de Ribeirão Preto foram encaminhados pelo Dr. Carlos Alberto Scrideli. 4.6 Avaliação da representatividade tumoral Para as amostras do HCB, um fragmento de tecido congelado de cada amostra selecionada para o estudo foi descalcificado, incluído em parafina e corado com hematoxilina/eosina, seguindo os protocolos padronizados no departamento de patologia do HCB. Em seguida, confeccionou-se lâminas de cada amostra e estas foram avaliadas pelo patologista Dr. Cristovam Scapulatempo Neto, para determinar a porcentagem de tumor presente no fragmento estocado e disponível no biobanco da instituição. As amostras provenientes da FMRP/USP foram submetidas a macrodissecção manual no laboratório de patologia da instituição, com o intuito de proporcionar o enriquecimento das amostras para células tumorais. A microdissecção das amostras foi realizada em criostato refrigerado e utilizando o composto Tissue Tek O.C.T (Sakura). Dois cortes de 810µm foram utilizados na macrodissecção, após a marcação da área tumoral pelo 17 patologista. A macrodissecção das amostras permite o enriquecimento maior do que 80% para células tumorais. 4.7 Extração de RNA A extração do RNA total de todas as amostras foi realizada a partir de um fragmento do tumor congelado, utilizando-se inicialmente o TRIZOL (Life Technologies), de acordo com as recomendações do fabricante. Brevemente, aproximadamente 30mg de tecido tumoral foram separadas em um tubo contendo 1mL de TRIZOL e esferas de cerâmica (CK28) para maceração. Os tecidos foram macerados utilizando o equipamento precellys 24 (Bertin Technologies) sendo submetidos a pelo menos 2 ciclos de maceração de 6500 rpm durante 10 segundos. Durante o intervalo de 5 minutos entre os ciclos, as amostras eram armazenadas no gelo. Após esse passo, todo o conteúdo foi transferido para um tubo de 1,5mL e acrescentou-se 200L de solução salina 0,9%. As amostras foram incubadas à temperatura ambiente por 5 minutos e adicionou-se 200L de clorofórmio. Em seguida, as amostras foram homogeneizadas com auxílio de vórtex por 15 segundos e centrifugadas a 12.000G por 15 minutos a 4°C. Após centrifugação, foi recolhido o sobrenadante (fase aquosa) contendo o RNA. O RNA foi precipitado acrescentando-se 500L de isopropanol gelado. Os tubos foram homogeneizados por inversão e armazenados no freezer a -20°C overnight. No dia seguinte, as amostras foram centrifugadas por 10 minutos a 12.000G na temperatura de 4°C. Em seguida, o sobrenadante foi descartado e realizaram-se duas lavagens do RNA com 1mL de etanol 75%, sendo os tubos centrifugados a 7.500G por 5 minutos na temperatura de 4°C. Ao final, descartou-se o sobrenadante, e o pellet de RNA resultante ficou à temperatura ambiente pelo tempo necessário para sua completa secagem. O RNA obtido foi ressuspendido com 20 µL de água milli-Q RNase-free. Para algumas amostras, que apresentaram baixa qualidade do RNA (RIN<6) a extração do RNA foi realizada novamente. Assim, o RNA foi extraído novamente utilizando-se o miRNeasy micro kit (Qiagen), de acordo com as recomendações do fabricante. Brevemente, 30mg de tecido tumoral foram adicionadas a 700µL de Qiazol nos tubos com esferas de cerâmica (CK28) para maceração. Os tecidos foram macerados da mesma maneira como descrito anteriormente, seguiram a extração pelo protocolo do kit e foram eluídas em 20 µL de água milli-Q RNase-free. 18 Após a extração, todas as amostras foram quantificadas e submetidas a análise de qualidade/integridade do RNA e permaneceram estocadas a -80°C até a sua utilização. 4.8 Avaliação da quantidade e da qualidade do RNA total A quantificação das amostras de RNA total foi realizada inicialmente no espectrofotômetro NanoVue® (GE Healthcare Life Sciences) por análise da absorbância a 260nm. A pureza do RNA extraído foi avaliada através do cálculo das razões A260/A280 e A260/A230, que indicam possíveis contaminações da amostra por proteínas e compostos fenólicos. Posteriormente, para garantir uma quantificação mais precisa das amostras, utilizamos uma metodologia de fluorescência com o equipamento Qubit (Life Technologies), usando um kit que possui sondas moleculares específicas de RNA e portanto, se houver proteína, DNA, ácidos nucleicos degradados presentes na amostra, estes não irão interferir na quantificação. A análise da qualidade do RNA total foi realizada por meio de eletroforese microfluida no equipamento Bioanalyzer 2100 (Agilent Technologies), utilizando o RNA Nano Chip kit (Agilent Technologies), de acordo com as recomendações do fabricante. Brevemente, após a preparação do chip foram adicionados 9µL de Small RNA conditioning solution na posição marcada como CS, 1µL de marcador na posição indicada como ladder e 5µL de RNA small marker em cada posição do chip reservada para as amostras, bem como na posição do marcador. As amostras foram desnaturadas por dois minutos a 70°C, para evitar a formação de estruturas secundárias do RNA, e foi adicionado 1µL (100ng) de cada amostra nas posições do chip marcadas de 1 a 12. O chip foi agitado no vórtex IKA MS3 (Manca), horizontalmente por um minuto a 2.200rpm e, em seguida, foi colocado no aparelho para leitura. Com o auxílio do Agilent 2100 Expert Software obteve-se o resultado (eletroferograma e densitometria dos géis) referente à qualidade das amostras que são representados pelo RNA Integrity Number (RIN). As amostras apresentando RIN entre 1 e 3 foram consideradas totalmente degradadas; entre 4-7 foram consideradas parcialmente degradadas; entre 8 e 10, amostras íntegras. 19 4.9. Análise de contaminação do RNA com DNA genômico Dos quatro genes escolhidos nesse estudo, dois (HOTAIR e BCYRN1) são formados por apenas um exon, sendo assim os ensaios (iniciadores e sonda) para RT-qPCR (PCR em tempo real) poderiam amplificar o DNA genômico, dessa forma prejudicando a análise de expressão destes genes. Assim, com o intuito de avaliar a presença de contaminação por DNA nas amostras de RNA previamente extraídas, nós realizamos uma PCR com iniciadores intrônicos para o gene KRAS em todas as amostras. A reação foi desenvolvida utilizando-se aproximadamente 100ng de RNA total, 1x Tampão GoTaq Flexi Buffer, 0,2mM de dNTPs, 2mM de MgCl2, 0,3µM dos iniciadores, 0,5U da enzima Go-Taq Hot Start Polymerase (Promega), em um volume final de 15µL. Os iniciadores utilizados foram: Direto - 5’ ATGTTCTAATATAGTCACATTTTC 3’ e Reverso - 5’ GTCCTGCACCAGTAATATGC 3’. As amostras foram incubadas no termociclador Veriti Thermal Cycler (Applied Biosystems), de acordo com as condições descritas na tabela 1. Tabela 1 - Condições de ciclagem para PCR do gene KRAS Estágio Temperatura Tempo Desnaturação inicial 96°C 10 minutos Desnaturação 96°C 45 segundos Anelamento 56,5°C 45 segundos Extensão 72°C 45 segundos Extensão final 72°C 10 minutos Final 4°C ∞ Ciclos 40 X A avaliação da amplificação do gene KRAS em todas as amostras foi realizada por eletroforese em gel de agarose 2% (Life Technologies), corado com corante Ez-vision in gel 10.000x (Amresco,USA). 4.10 Tratamento com DNase O tratamento com DNase (Sigma-Aldrich) foi realizado seguindo as orientações do fabricante. Brevemente, a partir de 1µg de RNA o tratamento foi realizado com 1x do 20 tampão da enzima, 1U de DNase I, em um volume final de 10µL. As amostras foram incubadas a 37°C por 20 minutos. Posteriormente, para inativação da enzima adicionou-se 1µL de Stop Solution (50mM EDTA) e as amostras foram incubadas a 70°C por 10 minutos, sendo em seguida colocadas no gelo. Para confirmar a eficiência do tratamento, após tratamento com DNase, a PCR do gene KRAS foi realizada novamente como descrito anteriormente (tópico 4.9). 4.11 Síntese de cDNA A síntese do cDNA foi realizada a partir de 1µg de RNA total tratado com DNAse utilizando o High Capacity cDNA Reverse Transcription Kit (Life Technologies), de acordo com as recomendações do fabricante. Brevemente, 1µg de RNA total tratado foi adicionado a 1x RT buffer, 4mM de dNTPs, 1x RT Random primers, 50U de MultiScribe Reverse Transcriptase e 20U de RNAse Inhibitor, em um volume final de 20µL. As amostras foram incubadas a 25°C, por 10 minutos, 37°C por 120 minutos, 85°C por 5 minutos. A reação foi realizada no Veriti Thermal Cycler (Applied Biosystems). 4.12 Análise de expressão dos RNAs não codificadores longos por RT-qPCR . A análise de expressão dos quatro lncRNAs foi realizada por RT-qPCR utilizando-se os ensaios PrimeTime qPCR assay (Integrated DNA Technologies). Estes ensaios contêm sondas e iniciadores específicos que permitem a amplificação e avaliação da expressão do gene de interesse. Após a síntese, 1µL do cDNA foi utilizado como molde nas reações de RT-qPCR que foram realizadas com 1x Kapa Probe Fast Master Mix (Kapa Biosystems), 1x PrimeTime qPCR assay (50X) específico para o gene em análise, em um volume final de 20µL. Essa reação foi desenvolvida a 95°C por 10 minutos; seguida de 40 ciclos a 95°C por 15 segundos e 60°C por 1 minuto. Além dos genes de interesse, foi realizada a avaliação dos genes endógenos ACTB e GAPDH que serviram para a normalização dos dados de expressão dos genes de interesse avaliados.A expressão normalizada de cada gene de interesse do estudo foi calculada por meio do modelo matemático 2-∆Ct, no qual o ∆Ct corresponde ao Ct do gene de interesse subtraído pelo Ct do gene normalizador81. A análise de expressão foi realizada no 7900 HT 21 Fast Real Time PCR System (Applied Biosystems) e todas as reações foram realizadas em duplicata. 4.13 Análise estatística Os dados obtidos dos prontuários foram digitados e arquivados em um banco de dados com auxílio do programa Statistical Package for Social Science (SPSS) versão 21.0. A análise estatística foi realizada utilizando-se o mesmo programa. Para este estudo, foram consideradas as variáveis epidemiológicas: sexo e idade ao diagnóstico e variáveis clínico-patológicas: tipo histológico, grau de Huvos, localização do tumor, tamanho do tumor, presença ou ausência de metástase e momento do diagnóstico da metástase. Para a análise estatística, algumas variáveis foram categorizadas da seguinte forma: Sexo: masculino ou feminino; Idade: menor/igual ou superior a 10 anos; Tipo histológico: osteoblástico ou condroblástico/fibroblástico/telangectásico; Grau de Huvos: grau 1/2 ou grau 3/4; Local do tumor: fêmur ou fíbula/tíbia/úmero; Tamanho do tumor: menor/igual ou superior a 12cm; a medida foi realizada por um radiologista através de exames de imagem ao diagnóstico (Raio X /Ressonância/Tomografia computadorizada; Presença de metástase: Ausente ou Presente; Momento do diagnóstico da metástase: não metastático ou metástase ao diagnóstico ou metástase após diagnóstico; As variáveis contínuas foram apresentadas como média, desvio padrão, mediana, valores mínimo e máximo. As variáveis categóricas foram apresentadas pelas frequências e porcentagens. Inicialmente, a expressão gênica foi considerada como uma variável numérica e a correlação da média de expressão dos genes com os fatores clínico-patológicos considerados no estudo foi realizada utilizando-se o teste de Mann-Whitney. A avaliação da diferença da qualidade do RNA medida pelo RIN, entre os métodos de extração utilizados (kit ou TRIZOL), também foi realizada pelo teste de Mann-Whitney. 22 Posteriormente, para categorizar a expressão dos genes foi determinado um ponto de corte utilizando a curva ROC e considerado o melhor ponto aquele que apresentou a melhor sensibilidade e especificidade na comparação entre pacientes não metastáticos e metastáticos. Para a avaliação desse ponto, foram analisados a área sob a curva (AUC), a sensibilidade, especificidade, valor preditivo positivo, valor preditivo negativo e acurácia e seus respectivos intervalos de confiança de 95%. Após essa análise, a expressão dos genes foi correlacionada com as características clínicas por meio do teste de qui-quadrado ou teste exato de Fisher, quando os pressupostos no teste do qui-quadrado não foram satisfeitos. Como a variável metástase foi divida em três categorias (não metastático, metástase ao diagnóstico ou metástase após o diagnóstico), utilizou-se o teste Kruskal-Wallis para avaliar a correlação com a expressão dos genes. A sobrevida global (SG) foi definida como o período da admissão (diagnóstico) até a avaliação mais recente de acompanhamento ou óbito. Para a análise da sobrevida livre de eventos (SLE), foi considerado como evento a recaída, a progressão de doença ou óbito (o que ocorreu primeiro). O tempo até o evento foi estimado entre a data do diagnóstico até a data do evento. Para estimar os tempos de sobrevida global e os tempos de sobrevida livre de evento utilizou-se o método de Kaplan-Meier. Para a comparação das curvas de sobrevida em relação a cada categoria dentro de uma determinada variável utilizou-se o teste nãoparamétrico de LogRank. Para todas as análises foi considerado um nível de significância de 5%. 23 5. 5.1 RESULTADOS Tamanho da Amostra Nos registros do HCB, das 175 amostras identificadas inicialmente, 76 permaneceram elegíveis para análise após avaliação dos prontuários. As demais foram excluídas pelos seguintes critérios: 68 amostras foram coletadas após início de quimioterapia; 12 amostras de pacientes que não receberam tratamento no Departamento de Pediatria do HCB; 05 amostras possuíam classificação incorreta pelo SisOnco; 03 amostras eram de recidiva; 02 amostras com radioterapia prévia; 03 amostras de metástase; 03 amostras com laudo inconclusivo; 02 amostras não estavam armazenadas no Biobanco; 01 amostra com fragmento pequeno, impossibilitando a avaliação pelo patologista. As 32 amostras de Ribeirão Preto já haviam sido avaliadas em relação a todos os critérios de inclusão e de exclusão com exceção da avaliação da integridade do RNA. Abaixo, segue uma representação esquemática da estratégia utilizada para a avaliação de expressão dos lncRNAs em osteossarcomas (figura 3). Cada etapa será descrita detalhadamente nos tópicos a seguir. 24 Figura 3 – Representação esquemática da estratégia desenvolvida neste trabalho para a avaliação da expressão de lncRNAs em osteossarcoma. 25 5.2 Avaliação da representatividade tumoral Inicialmente, as 76 amostras do HCB foram avaliadas para a confirmação da presença de tumor, das quais 56 foram confirmadas com pelo menos 50% de tecido tumoral no fragmento disponível. As 32 amostras provenientes da FMRP/USP já haviam passado por essa avaliação anteriormente e, após microdissecção manual houve um enriquecimento para 80% de tecido tumoral nestas amostras. Assim, foram consideradas para seguimento no projeto 88 amostras, das quais 71% (77 amostras) apresentaram pelo menos 80% de tecido tumoral viável. A figura 4 ilustra a quantidade de amostras analisadas e a porcentagem tumoral presente em cada uma delas. Figura 4. Representatividade tumoral nas amostras selecionadas para avaliação da expressão dos lncRNAs. Para as amostras do HCB esta avaliação foi realizada pelo patologista em lâmina de fragmento corado com hematoxilina e eosina. Para as amostra da FMRP/USP esta avaliação foi feita previamente e as amostras macrodissecadas garantindo a presença de pelo menos 80% de tecido tumoral viável. 5.3 Extração, quantificação e avaliação da qualidade do RNA total Inicialmente, a extração do RNA total foi realizada utilizando o método do TRIZOL. Após a extração, a quantificação e a pureza do RNA extraído foi avaliada pela análise de absorbância (260/280nm) utilizando o espectrofotômetro NanoVue. A avaliação da qualidade/integridade do RNA foi realizada com auxílio do equipamento Bioanalyzer 2100. Das 88 amostras avaliadas, três possuíam um fragmento tumoral muito pequeno que resultou em baixa quantidade de RNA e não foi possível obter o valor de RIN, e 26 nem a realização de nova extração/análise. Das 85 amostras restantes, 56 apresentaram degradação parcial do RNA (RIN < 6) e por isso, na tentativa de atingir melhores resultados, para 25 amostras com tecido remanescente o RNA foi extraído novamente utilizando-se o miRNeasy micro kit (Qiagen). A re-extração utilizando-se o kit não resultou em uma melhor qualidade do RNA visto que a maioria das amostras não apresentou diferença relevante em relação ao valor de RIN observado inicialmente. Neste processo de re-extração, apenas quatro amostras que inicialmente apresentaram RIN<4, foram recuperadas com RIN>4 e incorporadas na análise. A comparação dos valores do RIN entre os dois procedimentos de extração de RNA utilizados estão descritos na figura 5. Figura 5 - Comparação da integridade do RNA de amostras de osteossarcoma após extração com dois protocolos diferentes. O gráfico apresenta as médias e desvios padrão dos RINs obtidos nas extrações. A coluna da esquerda (em vermelho) representa as amostras extraídas com TRIZOL e a da direita (em azul), extraídas por kit. Não houve diferença estatística significativa (p=01642). Teste de Mann-Whitney. De acordo com o gráfico, podemos observar que não houve diferença significativa de qualidade do RNA entre os dois métodos de extração utilizados em nosso trabalho (p=0,1642). Em nosso trabalho, amostras com RIN > 4 apresentaram uma qualidade satisfatória para a análise, o que foi comprovado pela baixa variação dos Cts (1,27) observada na análise de expressão gênica dos genes normalizadores (endógenos), que será apresentada na tabela 4. Portanto, independentemente do método utilizado para a extração, as 31 amostras que obtiveram o RIN ≥ 4 foram selecionadas para o estudo. A mesma análise de qualidade foi aplicada as 32 amostras de RNA provenientes da FMRP/USP. Dessas, 12 amostras 27 apresentaram RIN > 4 e foram incluídas nas análises subsequentes. Assim, totalizando 43 amostras aptas a análise de expressão dos genes alvos (Figura 3). 5.4 Avaliação de contaminação com DNA genômico e tratamento das amostras de RNA com DNase Após a extração de RNA, para as amostras do HCB pôde-se observar que todas amplificaram o gene KRAS, o que confirma a contaminação por DNA nessas amostras (Figura 6). Figura 6 - Gel de agarose para a avaliação da amplificação do gene KRAS nas amostras de RNA. M: Ladder (1Kb plus - Life Technologies); C+: controle positivo (DNA genômico); C-: controle negativo (sem amostra). O tamanho esperado para o fragmento amplificado é de 202 pares de bases. Após o tratamento com DNAse, as amostras foram novamente submetidas a amplificação do gene KRAS, para a avaliação da eficiência do tratamento. O tratamento foi eficiente para eliminar a contaminação das amostras de RNA com DNA genômico, pois não observamos a amplificação do fragmento esperado deste gene, exceto para a amostra nº 70 (Figura 6). Como não havia mais RNA disponível dessa amostra, optamos por excluí-la das análises. Estes resultados foram obtidos após a quantificação do RNA com o aparelho Qubit, para os quais os motivos serão apresentados no tópico a seguir. Figura 7 - Gel de agarose para a avaliação da amplificação do gene KRAS nas amostras de RNA após tratamento com DNase. M: Ladder (1Kb plus - Life Technologies); L: linhagem celular de ostessarcoma SAOS; C+: controle positivo (DNA genômico); C- : controle negativo (sem amostra). O tamanho esperado para o fragmento amplificado é de 202 pares de bases. 28 5.5 Análise da expressão dos normalizadores e requantificação das amostras utilizando o Qubit Para a avaliação da expressão gênica dos alvos selecionados, utilizou-se a técnica de RT-qPCR. Inicialmente foi avaliada a expressão gênica dos dois genes endógenos (ACTB e GAPDH) com o intuito de selecionar o melhor gene normalizador, ou seja, aquele que apresentou menor variação de Cts dentre todas as amostras de osteossarcoma avaliadas. Sendo assim, a partir da quantificação obtida pelo Nanovue, 1µg de RNA foi tratado com DNase e após confirmação da eficiência do tratamento, as amostras foram submetidas a síntese de cDNA e avaliação da expressão dos genes endógenos. Essa análise revelou um alto desvio padrão na análise de expressão dos genes endógenos quando consideradas todas as amostras do estudo, o que não é satisfatório para genes que servirão de normalizadores para a análise de expressão gênica dos genes alvo (tabela 2). Tabela 2 – Média e desvio padrão dos Cts dos normalizadores ACTB e GAPDH, obtidos após análise de expressão gênica a partir de RNA quantificado por meio do Nanovue. Gene Média Ct Desvio Padrão ACTB 24,48 2,66 GAPDH 22,84 2,08 Devido à degradação parcial observada nas amostras de RNA (RIN entre 4 e 7), optouse por realizar a quantificação com o Qubit, já que esse método utiliza sondas moleculares específicas para a quantificação de RNA íntegro e portanto, proporciona uma análise mais fidedigna da quantidade de RNA nas amostras em estudo. Diante dos resultados de quantificação descritos na tabela 3, observa-se a grande variação apresentada entre a quantificação realizada pelo NanoVue e pelo Qubit, o que mostrou a necessidade de requantificar todas as amostras utilizando-se essa nova abordagem mais precisa. 29 Tabela 3 – Comparação da quantificação de RNA realizada pelos dois métodos utilizados neste trabalho Nº Amostra NanoVue (ng/µL) Qubit (ng/µL) 13 2292 17,4 48 1338 556 49 1325 794 63 305 119 64 4276 1350 Assim, após a requantificação das amostras utilizando-se o Qubit, duas foram excluídas devido à baixa quantidade de RNA disponível e impossibilidade de re-extração do RNA a partir do fragmento de tecido. Portanto, baseado na quantificação das amostras pelo Qubit, 1µg das 41 amostras de RNA remanescentes foi novamente tratado com DNase e após confirmação da eficiência do tratamento, houve a necessidade de exclusão de mais uma amostra. Dessa forma, o RNA de 40 amostras foi submetido a síntese de cDNA e avaliação da expressão dos genes endógenos. Ainda, na análise de expressão dos genes endógenos, quatro amostras apresentaram níveis muito baixos de expressão para estes genes (alto Ct) quando comparados a média geral das amostras. Mesmo sendo aprovadas em todos os parâmetros determinados em nosso trabalho, optamos por excluir essas amostras, pois isso poderia gerar um viés nas análises de expressão dos lncRNAs para estas amostras. Assim, as análises de expressão dos genes alvos seguiram com 36 amostras de RNAs de osteossarcoma. A análise de expressão dos genes endógenos (ACTB e GAPDH), nestas 36 amostras revelou que o gene ACTB apresentou a menor variação de expressão entre todas amostras avaliadas, sendo então escolhido para normalização dos dados de expressão gênica dos genes alvos (Tabela 4). Tabela 4 - Média e desvio padrão dos Cts dos normalizadores ACTB e GAPDH, obtidos após análise de expressão gênica a partir de RNA quantificado por meio do Qubit. Gene Média Ct Desvio Padrão ACTB 19,50 1,27 GAPDH 19,00 2,06 30 5.6 Características clínicas, patológicas e demográficas dos pacientes com osteossarcoma A caracterização das amostras foi realizada para os 36 pacientes, para os quais a análise de expressão dos genes foi realizada. A maioria eram do sexo feminino (55,6%), com idade entre 4 e 29 anos. A média de idade ao diagnóstico foi de 15 anos. Quanto às características clínicas, ao diagnóstico, a maioria dos pacientes (53,6%) apresentaram tumores menos volumosos (<12cm). Dos 36 pacientes do estudo, foi possível obter o tamanho do tumor de 20 pacientes, sendo que a medida de 9 pacientes foi obtida através da radiografia convencional, 7 através de ressonância magnética e 4 por tomografia computadorizada. A localização mais frequente do tumor foi o fêmur em 16 pacientes (44,4%), sendo o subtipo histológico mais comum o osteoblástico, que foi confirmado em 28 casos (77,8%). Em relação ao grau de resposta à quimioterapia (grau de Huvos-Ayala), a maioria dos pacientes apresentou resposta parcial com mais de 50% de necrose, grau 2 de Huvos-Ayala que foi observado em 14 pacientes (41,17%), sendo que 12 pacientes (35,3%) não responderam a quimioterapia (grau 1 de Huvos-Ayala), seis (17,64%) apresentaram mais que 90% de necrose tumoral, com tumor viável presente, dois (5,88%) apresentaram grau 4, sem tumor viável e, para dois pacientes o grau de Huvos não se aplicava pois os pacientes não realizaram cirurgia após quimioterapia. Em relação à presença de metástases, 20 pacientes (55,6%) não apresentaram metástases, e nos 16 pacientes metastáticos (44,4%), a metástase pulmonar foi a mais frequente. Todos esses dados podem ser visualizados na tabela 5. 31 Tabela 5 – Características clínicas, patológicas e demográficas dos pacientes com osteossarcoma para a análise de expressão dos lncRNAs. Variável Sexo Idade ao diagnóstico Local do tumor Tipo histológico Grau de Huvos* Tamanho do tumor* Metástases* Categoria n (%) feminino 20 (55,6) masculino 16 (44,4) <10 anos 7 (19,4) >10 anos 29 (80,6) Média 15 anos Fêmur 16 (44,4) Tíbia 11 (30,6) Úmero 6 (16,7) Fíbula 3 (8,3) Osteoblástico 28 (77,8) Condroblástico 5 (13,9) Fibroblástico 2 (5,6) Telangectásico 1 (2,8) Grau 1 12 (35,3) Grau 2 14 (41,17) Grau 3 6 (17,64) Grau 4 2 (5,88) <12 15 (53,6) >=12 13 (46,4) Não metastático 20 (57,1) Metástase ao diagnóstico 7 (20,0) Metástase após diagnóstico 8 (22,9) *nº de pacientes cuja variável foi possível obter os dados 5.7 Análise da expressão dos lncRNAs O presente estudo avaliou a expressão dos lncRNAs nas 36 amostras tumorais de OS por meio da RT-qPCR. Os resultados obtidos e normalizados mostram uma ampla variação na expressão dos quatro genes avaliados. Interessante notarmos que para algumas amostras, a expressão dos 32 genes não foi detectada demonstrando que os genes não eram expressos nestas amostras, principalmente para os genes HOTAIR e BCYRN1. Neste experimento, foi utilizada uma linhagem celular de osteossarcoma (SAOS2) a fim de verificar o perfil de expressão desses genes nesta linhagem. Como podemos notar, estes genes apresentam uma baixa expressão nessa linhagem celular de osteossarcoma. Abaixo estão demonstrados os gráficos (Figura 8), referentes a expressão de cada um dos lncRNAs nas 36 amostras de OS. Figura 8 - Expressão normalizada dos lncRNAs em osteossarcoma avaliada por RT-qPCR. A: MALAT1; B: HULC; C: HOTAIR; D: BCYRN1. No eixo x, cada número representa uma amostra. No eixo y está representado o valor normalizado de expressão do lncRNA avaliado. A normalização foi realizada de acordo com o modelo matemático 2- ΔCt, sendo o gene endógeno ACTB utilizado como normalizador. 33 Figura 8 (Continuação) - Expressão normalizada dos lncRNAs em osteossarcoma avaliada por RT-qPCR. A: MALAT1; B: HULC; C: HOTAIR; D: BCYRN1. No eixo x, cada número representa uma amostra. No eixo y está representado o valor normalizado de expressão do lncRNA avaliado. A normalização foi realizada de acordo com o modelo matemático 2- ΔCt, sendo o gene endógeno ACTB utilizado como normalizador. 5.8 Associação da expressão dos lncRNAs com os fatores clínico-patológicos dos pacientes Tendo em vista o potencial papel dos lncRNAs na carcinogênese e sua influência no prognóstico de pacientes com câncer, foi avaliada a correlação da expressão desses genes com os fatores clínicos-patológicos considerados no estudo como tipo histológico, grau de Huvos, sexo, idade ao diagnóstico, local do tumor, tamanho do tumor e presença de metástase. 34 Considerando a expressão dos genes como uma variável numérica, podemos observar que para o gene BCYRN1 sua maior expressão está associada significativamente com pacientes que apresentam idade acima de 10 anos (p=0,036). Apesar de não significativos, nós podemos destacar algumas observações como por exemplo o grupo de tumores com grau histológico condroblástico, fibroblástico e telangectásico, apresenta em média pelo menos quatro vezes maior expressão de BCYRN1 quando comparado aos tumores osteoblásticos. Além disso, a maior expressão deste gene também pode ser observada nos tumores menos responsivos a quimioterapia (grau Huvos 1/2) quando comparados aos que respondem (Huvos 3/4). Em relação a presença de mestástases, tumores associados a metástases apresentaram maior expressão de BCYRN1 (em média três vezes maior) em comparação aos casos não metastáticos (Tabela 6). Para o gene HOTAIR, apesar da associação da expressão gênica e as características clínico-patológicas dos pacientes não serem significativas, podemos observar maior expressão de HOTAIR no grupo de tumores com tipo histológico condroblástico, fibroblástico e telangectásico, aproximadamente 13 vezes maior, quando comparado aos tumores osteoblásticos. Podemos observar também uma maior expressão de HOTAIR nas amostras de pacientes não metastáticos, ainda em tumores localizados na fíbula, tíbia ou úmero ou que responderam pior à QT pré-operatória (Huvos 1/2) e que ao diagnóstico apresentaram tamanho do tumor menor que 12cm (Tabela 7). Para o gene HULC, a associação da expressão gênica e as características clínicopatológicas dos pacientes também não foram significativas, entretanto podemos observar que a média de expressão foi maior em pacientes que apresentaram tumores do tipo histológico osteoblástico, em tumores de pacientes com idade igual ou superior a 10 anos e em tumores para os quais o paciente apresentou metástase após o diagnóstico (Tabela 8). Em relação ao MALAT1, sua maior expressão foi associada significativamente a pacientes com idade igual ou superior a 10 anos. Apesar de não encontrarmos associação significativa às demais características clínico-patológicas, observamos a maior expressão de MALAT1 em pacientes do sexo masculino, em tumores localizados na fíbula, tíbia ou úmero. Ainda, quando consideramos a presença de metástase independente do momento, observamos uma expressão um pouco aumentada de MALAT1 em pacientes não metastáticos. Entretanto, quando os pacientes que tem metástase são estratificados em 35 dois grupos, observamos que a maior expressão do gene ocorre nos tumores de pacientes que desenvolvem metástase ao longo do tratamento (Tabela 9). Analisando os resultados obtidos, podemos notar que as amostras analisadas mostraram uma ampla variação de expressão dos quatro genes analisados, como demonstrado anteriormente (Figura 8). Os valores de média, desvio padrão, mediana e percentis dos resultados obtidos para os genes analisados estão demonstrados nas tabelas de 6 a 9. 36 Tabela 6 – Comparação da média de expressão do BCYRN1 com as características clínico-patológicas dos pacientes avaliados. Contagem Média Desvio padrão Mínimo Osteoblástico 28 0,781883 0,828815 0,053557 0,209929 0,482505 1,111098 3,509397 Condroblástico/Fibroblástico Telangectásico 8 3,389459 5,869342 0,081406 0,490397 1,275666 2,941804 17,618533 Grau 1 / 2 26 1,581165 3,385022 0,061554 0,252257 0,869527 1,426408 17,618533 Grau 3 / 4 8 0,509721 0,432008 0,053557 0,155135 0,408658 0,798371 1,299885 Feminino 20 1,865601 3,865963 0,053557 0,295323 0,815225 1,284110 17,618533 Masculino 16 0,731024 0,662778 0,081406 0,185287 0,428737 1,319098 2,160619 <10 anos >=10 anos 7 29 0,675498 1,526894 1,347527 3,201655 0,053557 0,061554 0,081406 0,351490 0,190994 0,864022 0,302513 1,299885 3,722989 17,618533 Fêmur 16 1,036519 1,100962 0,061554 0,327002 0,679728 1,284110 3,722989 Fíbula, Tíbia e Úmero 20 1,621205 3,844194 0,053557 0,185287 0,640261 1,295311 17,618533 <12 cm 15 1,102877 1,078174 0,053557 0,106236 0,864022 1,465697 3,722989 >=12 cm 13 0,911535 0,944459 0,061554 0,302513 0,450207 1,299885 3,509397 Ausentes 20 0,687126 0,565351 0,053557 0,240560 0,553916 0,951154 2,160619 Presentes 16 2,204118 4,285407 0,061554 0,136692 0,999978 2,178777 17,618533 Não metastático 20 0,68742 0,56535 0,5356 0,24087 0,55397 0,95125 2,16062 Metástase ao diagnostico* 7 1,423940 1,56189 0,6155 0,10624 0,87504 3,51087 3,73304 Metástase após diagnostico* 8 p-valor < 0,05 *nº de pacientes cuja variável foi possível obter os dados 2,999230 6,0173 0,8144 0,12866 0,78793 2,18982 17,69960 Tipo histológico Grau de Huvos* Percentil 25 Mediana Percentil 75 Máximo Sexo Idade ao diagnóstico Local do tumor Tamanho do tumor* 0,138 0,155 0,504 0,036 0,610 Metástase Metástase* p-valor 0,695 0,373 0,787 37 Tabela 7 - Comparação da média de expressão do HOTAIR com as características clínico-patológicas dos pacientes avaliados. Tipo histológico Grau de Huvos* Sexo Idade ao diagnóstico Local do tumor Contagem Média Desvio padrão Mínimo Percentil 25 Mediana Percentil 75 Máximo Osteoblástico 28 0,004895 0,010908 0,000006 0,000200 0,000649 0,003271 0,049928 Condroblástico/Fibroblástico Telangectásico 8 0,053119 0,144645 0,000001 0,000248 0,002224 0,004468 0,411069 Grau 1 / 2 26 0,020308 0,080410 0,000001 0,000343 0,000981 0,004132 0,411069 Grau 3 / 4 8 0,003746 0,007745 0,000074 0,000118 0,000271 0,003271 0,022579 Feminino 20 0,004219 0,007400 0,000001 0,000292 0,000649 0,004029 0,025486 Masculino 16 0,029852 0,102391 0,000084 0,000184 0,000981 0,002734 0,411069 <10 anos >=10 anos 7 29 0,001623 0,018988 0,001998 0,076161 0,000074 0,000001 0,000153 0,000299 0,000535 0,000857 0,003926 0,003902 0,004902 0,411069 Fêmur 16 0,006980 0,013925 0,000006 0,000321 0,000649 0,003914 0,049928 Fíbula, Tíbia e Úmero 20 0,022517 0,091503 0,000001 0,000161 0,001088 0,003481 0,411069 <12 cm 15 0,029670 0,105558 0,000130 0,000385 0,001613 0,003926 0,411069 >=12 cm 13 0,004059 0,008893 0,000006 0,000167 0,000343 0,001106 0,025486 Ausentes 20 0,023479 0,091391 0,000074 0,000184 0,000785 0,004017 0,411069 Presentes 16 0,005777 0,013314 0,000001 0,000249 0,000936 0,002772 0,049928 Não metastático 20 0,023573 0,091767 0,000077 0,000185 0,000785 0,004040 0,412760 Metástase ao diagnostico 7 0,004605 0,009369 0,000028 0,000205 0,000555 0,003925 0,025625 Metástase após diagnostico 8 0,007505 0,017220 0,000001 0,000279 0,001455 0,003315 0,049940 0,543 Tamanho do tumor* Metástase Metástase* p-valor 0,256 0,874 0,510 0,874 0,093 p-valor < 0,05 *nº de pacientes cuja variável foi possível obter os dados 0,799 0,966 38 Tabela 8 - Comparação da média de expressão do HULC com as características clínico-patológicas dos pacientes avaliados. Desvio padrão Contagem Média Osteoblástico 28 0,000038420 0,000036056 0,000000999 0,000009198 0,000026801 0,000062379 0,000126349 Tipo histológico Condroblástico/Fibroblástico/ Telangectásico 8 0,000021071 0,000037211 0,000000904 0,000001697 0,000006115 0,000020671 0,000110694 Grau de Huvos* Grau 1 / 2 26 0,000032895 0,000035429 0,000000904 0,000006021 0,000019150 0,000058980 0,000119445 Grau 3 / 4 8 0,000047261 0,000041736 0,000001921 0,000008204 0,000051891 0,000064813 0,000126349 Feminino Masculino <10 anos 20 16 7 0,000031637 0,000038224 0,000012347 0,000036392 0,000000999 0,000002119 0,000013231 0,000053670 0,000119445 0,000037522 0,000000904 0,000006996 0,000025021 0,000067190 0,000126349 0,000015860 0,000001921 0,000002102 0,000009950 0,000012230 0,000046800 >=10 anos 29 0,000039927 0,000038205 0,000000904 0,000007970 0,000025805 0,000064756 0,000126349 Fêmur 16 0,000032168 0,000034274 0,000000904 0,000003990 0,000017751 0,000057981 0,000119445 Fíbula, Tíbia e Úmero 20 0,000036482 0,000038988 <12 cm 15 0,000028590 0,000030117 0,000000999 0,000001960 0,000017106 0,000056982 0,000087958 >=12 cm Ausentes 13 20 0,000044040 0,000036495 0,000041784 0,000000904 0,000010426 0,000033252 0,000060002 0,000126349 0,000031211 0,000001018 0,000012193 0,000026801 0,000057981 0,000126349 Presentes 16 0,000032151 0,000043198 0,000000904 0,000001626 0,000007986 0,000069012 0,000119445 Não metastático 20 0,00003 0,00003 0,00000 0,00001 0,00003 0,00005 0,00013 Metástase ao diagnostico 7 0,00002 0,00004 0,00000 0,00000 0,00001 0,00002 0,00012 Metástase após diagnostico 8 0,00005 0,00005 0,00000 0,00000 0,00003 0,00010 0,00012 Sexo Idade ao diagnóstico Local do tumor Tamanho do tumor* Metástase Metástase* p-valor < 0,05 *nº de pacientes cuja variável foi possível obter os dados Mínimo Percentil 25 Mediana Percentil 75 Máximo 0,000000999 0,000005125 0,000020671 0,000067190 0,000126349 p-valor 0,119 0,394 0,484 0,105 0,702 0,369 0,171 0,378 39 Tabela 9 - Comparação da média de expressão do MALAT1 com as características clínico-patológicas dos pacientes avaliados. Contagem Média 28 4,49462 Osteoblástico Tipo histológico Grau de Huvos* Sexo Idade ao diagnóstico Local do tumor Tamanho do tumor* Metástase Metástase* Desvio padrão Mínimo Percentil 25 Mediana Percentil 75 Máximo p-valor 2,59922 0,31235 2,67482 3,83030 5,87172 10,31942 0,879 Condroblástico/Fibroblástico Telangectásico 8 7,11100 7,97581 1,11040 1,62371 3,56961 12,57206 20,24685 Grau 1 / 2 26 5,26356 4,90580 0,31235 2,53629 3,83030 5,83639 20,24685 Grau 3 / 4 8 4,92796 2,93913 1,82188 2,51630 4,12311 7,00178 10,31942 Feminino 20 4,47554 4,14551 0,31235 2,45454 3,33675 4,66701 19,30773 Masculino 16 5,82666 4,67299 1,42553 3,25912 3,88384 7,59785 20,24685 <10 anos 7 3,36515 2,95060 1,11040 1,82188 2,53752 2,87323 9,90586 >=10 anos 29 5,48901 4,60057 0,31235 2,93827 3,98345 6,67541 20,24685 Fêmur 16 3,97204 2,60823 0,31235 2,22625 3,29498 5,43569 9,78187 Fíbula, Tíbia e Úmero 20 5,95924 5,29724 1,11040 2,87120 3,94474 6,62821 20,24685 <12 cm 15 3,80605 2,55096 0,31235 2,41400 2,93827 5,06803 10,31942 >=12 cm 13 4,27727 2,33912 1,42553 2,93028 3,79895 4,05027 9,78187 Ausentes 20 5,67148 4,29679 1,95742 3,20004 3,94474 7,00178 20,24685 Presentes 16 4,33174 4,49858 0,31235 1,82685 2,93428 4,66701 19,30773 Não metastático 20 5,68325 4,30610 1,95837 3,20250 3,95382 7,02332 20,28665 Metástase ao diagnostico 7 2,60455 1,23704 0,31453 1,83184 2,87658 3,65196 4,03885 8 6,21568 5,81494 1,11045 2,37683 4,67390 7,59983 19,31390 Metástase após diagnostico p-valor < 0,05 *nº de pacientes cuja variável foi possível obter os dados 0,685 0,252 0,048 0,214 0,534 0,126 0,100 40 5.9 Associação do perfil de expressão dos lncRNAs com as características clínico- patológicas dos pacientes Com o intuito de categorizar a expressão dos genes (maior e menor expressão) nós utilizamos a análise da curva ROC. Uma vez que esses genes são marcadores de metástase em outros tumores, o ponto determinado na curva ROC para cada gene foi aquele que apresentou a melhor especificidade e sensibilidade para discriminar entre pacientes metastáticos e não metastáticos. Assim, considerando os resultados de expressão obtidos para cada gene, podemos observar que a análise da curva ROC não revelou nenhum ponto de corte com significância estatística que permitisse a discriminação das amostras entre metastáticas e não metastáticas (tabela 10 e 11) Sendo assim, utilizamos para cada gene o ponto da curva ROC que apresentava a maior sensibilidade e especificidade para determinação desses grupos. Portanto, para as amostras cujos resultados de expressão encontravam-se acima deste ponto foram consideradas com alta expressão do gene, em contrapartida àquelas abaixo do ponto determinado consideradas com baixa expressão do gene. Tabela 10 – Área sob a curva, desvio padrão, p-valor e intervalo de confiança determinados pela curva ROC. IC 95% Gene Área sob a curva Desvio padrão p-valor Limite inferior Limite superior BCYRN1 0,588 0,105 0,373 0,382 0,793 HOTAIR 0,475 0,099 0,799 0,281 0,669 HULC 0,366 0,106 0,171 0,158 0,573 MALAT1 0,65 0,096 0,126 0,463 0,837 41 Tabela 11 – Ponto de corte, sensibilidade, especificidade, valor preditivo positivo (VPP), valor preditivo negativo (VPN) e acurácia, de acordo com a curva ROC. Gene Ponto de corte Sensibilidade % Especificidade % VPP VPN Acurácia BCYRN1 0,86952666 40 (19,17-63,93) 35,71 (18,67-55,93) 30,77 (14,37-51,79) 45,45 (24,42-67,77) 37,5 (25,22-51,64) HOTAIR 0,00056915 50 (27,23-72,77) 67,9 (47,65-84,09) 52,6 (28,9-75,51) 65,5 (45,67-82,04) 60,4 (46,31-72,98) 0,0000107873 55 (31,55-76,9) 71,4 (51,33-86,74) 57,9 (33,53-79,7) 69 (49,17-84,68) 64,6 (50,44-76,57) 3,6699867 60 (36,07-80,83) 71,4 (51,33-86,74) 60 (36,07-80,83) 71,4 (51,33-86,74) 66,7 (52,54-78,32) HULC MALAT1 Assim, após a categorização da expressão dos genes nas amostras avaliadas, nós realizamos as análises de associação do padrão de expressão dos genes com as características clínico-patológicas dos pacientes. Para o gene BCYRN1, nossos resultados mostraram que 90,5% dos pacientes que apresentaram baixa expressão do gene pertenciam ao tipo histológico osteoblástico (p=0,046). Apesar de não ter significância estatística, podemos observar que dos pacientes que apresentaram alta expressão do gene 93,3% tinham idade ao diagnóstico maior ou igual a 10 anos e 93% apresentaram baixa resposta ao tratamento (Grau Huvos 1/2). Ainda, daqueles pacientes cuja expressão foi alta, 60,0% apresentavam metástase em algum momento (Tabela 12). Em relação ao HOTAIR, 75% dos pacientes com alta expressão do gene apresentaram tamanho do tumor < 12cm (p=0,049) e, apesar de não significativo, considerando a baixa expressão, 60% não eram metastáticos (Tabela 13). Para o gene HULC não houve associação estatística significativa entre a o perfil de expressão do gene e as características clínico-patológicas avaliadas. Entretanto, podemos destacar algumas observações, como por exemplo 88,9% dos pacientes que apresentaram alta expressão do gene pertencia ao tipo histológico osteoblástico e, 94,4% apresentavam idade ao diagnóstico igual ou superior a 10 anos (p-valor = 0,088) (Tabela 14). Em relação a expressão do MALAT1, 95,5% dos pacientes que apresentaram alta expressão tinham 10 anos ou mais ao diagnóstico (p=0,008). Apesar dos outros resultados encontrados não terem sido estatisticamente significativos, podemos destacar que a maioria 42 dos pacientes (68,2%) com alta expressão do gene não eram metastáticos e a baixa expressão do gene foi vista no grupo de pacientes do sexo feminino (71,4%) e que apresentavam tamanho do tumor ao diagnóstico menor do que 12 cm (66,7%). (Tabela 15). 43 Tabela 12 – Associação do perfil de expressão do BCYRN1 com as características epidemiológicas e clínicopatológicas. Baixa expressão Osteoblástico Tipo histológico Grau de Huvos* Sexo Local do tumor Idade ao diagnóstico Tamanho do tumor* Metástase Metástase* Alta expressão p-valor n % n % 19 90,5 9 60,0 0,046 Condroblástico/Fibroblástico/ Telangectásico 2 9,5 6 40,0 Grau 1 / 2 13 65,0 13 92,9 Grau 3 / 4 7 35,0 1 7,1 Feminino 11 52,4 9 60,0 Masculino 10 47,6 6 40,0 Fêmur 9 42,8 7 46,7 Fíbula, Tíbia e Úmero 12 57,2 8 53,3 <10 anos 6 28,6 1 6,7 >=10 anos 15 71,4 14 93,3 <12 cm 8 50,0 7 58,3 >=12 cm 8 50,0 5 41,7 Ausentes 14 66,7 6 40,0 Presentes 7 33,3 9 60,0 Não metastático 14 66,7 6 42,9 Metástase ao diagnóstico 3 14,3 4 28,6 Metástase após diagnóstico 4 19 4 28,6 0,102 0,650 0,821 0,200 0,662 0,112 0,410 44 Tabela 13 – Associação do perfil de expressão do HOTAIR com as características epidemiológicas e clínicopatológicas Baixa expressão Alta expressão Tipo histológico Grau de Huvos* Sexo Local do tumor Idade ao diagnóstico Tamanho do tumor* Metástase Metástase* n % n % Osteoblástico 17 85,0 11 68,8 Condroblástico/Fibroblástico/ Telangectásico 3 15,0 5 31,3 Grau 1 / 2 14 73,7 12 80,0 Grau 3 / 4 5 26,3 3 20,0 feminino 11 55,0 9 56,3 masculino 9 45,0 7 43,8 Fêmur 10 50,0 6 37,5 Fíbula, Tíbia e Úmero 10 50,0 10 62,5 <10 anos 4 20,0 3 18,8 >=10 anos 16 80,0 13 81,3 <12 cm 6 37,5 9 75,0 >=12 cm 10 62,5 3 25,0 Ausentes 12 60,0 8 50,0 Presentes 8 40,0 8 50,0 Não metastático 12 63,2 8 50,0 Metástase ao diagnóstico 4 21,1 3 18,8 Metástase após diagnóstico 3 15,8 5 31,3 p-valor 0,422 0,999 0,940 0,453 0,999 0,049 0,549 0,585 45 Tabela 14 – Associação do perfil de expressão do HULC com as características epidemiológicas e clínicopatológicas HULC Baixa expressão Tipo histológico Grau de Huvos* Sexo Local do tumor Idade ao diagnóstico Tamanho do tumor* Metástase Metástase* Alta expressão n % n % Osteoblástico 12 66,7 16 88,9 Condroblástico/Fibroblástico/ Telangectásico 6 33,3 2 11,1 Grau 1 / 2 13 81,3 13 72,2 Grau 3 / 4 3 18,8 5 27,8 Feminino 11 61,1 9 50,0 Masculino 7 38,9 9 50,0 Fêmur 8 44,4 8 44,4 Fíbula, Tíbia e Úmero 10 55,6 10 55,6 <10 anos 6 33,3 1 5,6 >=10 anos 12 66,7 17 94,4 <12 cm 8 61,5 7 46,7 >=12 cm 5 38,5 8 53,3 Ausentes 8 44,4 12 66,7 Presentes 10 55,6 6 33,3 Não metastático 8 47,1 12 66,7 Metástase ao diagnóstico 5 29,4 2 11,1 Metástase após diagnóstico 4 23,5 4 22,2 p-valor 0,228 0,693 0,502 0,999 0,088 0,431 0,18 0,384 46 Tabela 15 – Associação do perfil de expressão do MALAT1 com as características epidemiológicas e clínicopatológicas Tipo histológico Grau de Huvos* Sexo Local do tumor Baixa expressão n % Alta expressão n % Osteoblástico 10 71,4 18 81,8 Condroblástico / Fibroblástico / Telangectásico 4 28,6 4 18,2 Grau 1 / 2 10 76,9 16 76,2 Grau 3 / 4 3 23,1 5 23,8 Feminino 10 71,4 10 45,5 Masculino 4 28,6 12 54,5 Fêmur 8 57,1 8 36,4 Fíbula, Tíbia e Úmero 6 42,9 14 63,6 Idade ao diagnóstico <10 anos 6 42,9 1 4,5 >=10 anos 8 57,1 21 95,5 Tamanho do tumor* <12 cm 8 66,7 7 43,8 >=12 cm 4 33,3 9 56,3 Ausentes 5 35,7 15 68,2 Presentes 9 64,3 7 31,8 Não metastático 5 38,5 15 68,2 Metástase ao diagnóstico 5 38,5 2 9,1 Metástase após diagnóstico 3 23,1 5 22,7 Metástase Metástase* p-valor 0,683 0,999 0,126 0,221 0,008 0,229 0,056 0,101 47 5.10 Análise de sobrevida global e sobrevida livre de eventos A população avaliada em nosso estudo sem metástase ao diagnóstico e com metástase ao diagnóstico apresenta sobrevida global em 5 anos de 62,7% e 28,6%, respectivamente e sobrevida livre de eventos de 59,6 e 28,6%, respectivamente (figura 9). Figura 9 - Análise de sobrevida global e sobrevida livre de eventos para os pacientes metastáticos e não metastáticos ao diagnóstico tratados pelo protocolo GLATO 2006 no Departamento de Pediatria do Hospital de Câncer de Barretos no período de 2006 a 2013. Em relação às características epidemiológicas, os resultados encontrados não foram estatisticamente significativos para a análise de sobrevida, no entanto, podemos observar que tanto a sobrevida global como a sobrevida livre de eventos em 5 anos é maior em pacientes do sexo masculino (Tabela 16 e 17). Tabela 16 - Sobrevida global (SG) em 1 ano, 3 anos e 5 anos em relação as características epidemiológicas. Variável Sexo Idade Categoria Feminino SG 1 ano 73,7 3 anos 43,6 5 anos 43,6 Masculino < 10 anos > 10 anos 93,3 85,7 81,5 79 57,1 60,9 64,6 57,1 51,5 p-valor 0,101 0,885 48 Tabela 17 - Sobrevida livre de eventos (SLE) em 1 ano, 3 anos e 5 anos em relação as características epidemiológicas. SLE Variável Categoria 1 ano 3 anos 5 anos p-valor Sexo Idade Feminino 60 42,0 42,0 Masculino < 10 anos > 10 anos 93,3 71,4 75,0 66,7 57,1 51,6 66,7 57,1 51,6 0,136 0,912 Quanto às características clínico-patológicas, não encontramos significância estatística em nenhuma das variáveis analisadas em relação a análise de sobrevida, mas podemos observar que tanto a SG como a SLE em 5 anos é maior para os pacientes com tipo histológico osteoblástico, que responderam melhor à quimioterapia (Grau de Huvos 3/4), nos tumores de localização tíbia/fíbula/úmero (Tabela 18 e 19). Tabela 18 - Sobrevida global (SG) em 1 ano, 3 anos e 5 anos em relação as características clínico-patológicas. Variável SG Categoria Osteoblástico 1 ano 3 anos 5 anos p-valor 82,1 66,2 57 Tipo histológico 0,225 Condroblástico/Fibroblástico/ 83,3 Telangectásico Grau de Huvos Local do Tumor Tamanho do tumor 33,3 33,3 Grau 1/2 83,3 56,7 46,4 Grau 3/4 87,5 72,9 72,9 Fêmur 60 52,5 39,4 Tíbia /Fíbula / Úmero 94,7 68,4 63,2 < 12 cm 86,7 47,7 47,7 > 12 cm 66,7 66,7 55,6 0,287 0,063 0,785 49 Tabela 19 - Sobrevida livre de eventos (SLE) em 1 ano, 3 anos e 5 anos em relação as características clínicopatológicas. Variável SLE Categoria 1 ano 3 anos 5 anos p-valor Osteoblástico 78,6 59,2 59,2 Tipo histológico 0,1 Condroblástico/Fibroblástico/ 57,1 Telangectásico Grau de Huvos Local do tumor Tamanho do tumor 28,6 28,6 Grau 1/2 72 46,9 46,9 Grau 3/4 87,5 72,9 72,9 Fêmur 53,3 44,4 44,4 Tíbia /Fíbula / Úmero 90 60 60 < 12 cm 73,3 49,5 49,5 > 12 cm 66,7 58,3 58,3 0,273 0,150 0,759 Em relação a expressão dos genes, também não observamos um impacto significativo na sobrevida global e sobrevida livre de eventos. Entretanto, podemos observar que os pacientes que apresentaram maior expressão dos genes BCYRN1 e HOTAIR, apresentaram uma menor sobrevida global e sobrevida livre de eventos em 5 anos (tabela 20 e 21; figura 10 e 11). Tabela 20 - Sobrevida global (SG) em 1 ano, 3 anos e 5 anos em relação a expressão dos lncRNAs. Variável Categoria SG 1 ano 3 anos 5 anos p-valor Baixa expressão 81 70,8 59,4 Alta expressão 84,6 40,4 40,4 Baixa expressão 83,3 64,8 64,8 Alta expressão 81,3 54,2 40,6 Baixa expressão 81,3 60,9 60,9 BCYRN1 0,295 HOTAIR 0,242 HULC 0,525 Alta expressão 83,3 58,9 44,9 Baixa expressão 84,6 59,8 49,9 Alta expressão 59,,9 54,5 MALAT1 0,778 81 50 Tabela 21 - Sobrevida livre de eventos (SLE) em 1 ano, 3 anos e 5 anos em relação a expressão dos lncRNAs. Variável Categoria SLE 1 ano 3 anos 5 anos p-valor Baixa expressão 76,2 61,0 61,0 Alta expressão 71,4 40,2 40,2 Baixa expressão 78,9 62,3 62,3 Alta expressão 68,8 41,3 41,3 Baixa expressão 76,5 57,8 57,8 BCYRN1 0,313 HOTAIR 0,224 HULC 0,557 Alta expressão 72,2 48,1 48,1 Baixa expressão 69,2 53,8 53,8 Alta expressão 53,1 53,1 MALAT1 0,753 77,3 51 Figura 10 - Análise de sobrevida global de acordo com o perfil de expressão dos lncRNAs. (A) BCYRN1; (B) HOTAIR; (C) HULC; (D) MALAT1. 52 Figura 11 - Análise de sobrevida livre de eventos de acordo com o perfil de expressão dos lncRNAs. (A) BCYRN1; (B) HOTAIR; (C) HULC; (D) MALAT1. 53 6 6.1 DISCUSSÃO Otimização do protocolo de extração e análise da qualidade do RNA Na otimização do protocolo de extração e análise da qualidade do RNA, observamos aspectos importantes durante esses passos que muitas vezes não são descritos na literatura, ou simplesmente não realizados com o rigor necessário. A avaliação do patologista para garantir uma boa representatividade tumoral foi uma etapa crucial, pois a contaminação das amostras tumorais com células normais pode levar a uma interpretação errônea das análises de expressão dos genes. Após essa análise para confirmação da representatividade tumoral das nossas amostras, 20 foram excluídas do estudo por possuírem quantidade menor que 50% de tumor, o que ainda é uma quantidade razoável de células tumorais. Em uma análise de correlação entre a pureza do tecido tumoral e a expressão gênica foi demonstrado que amostras com menos do que 50% de tecido tumoral apresentam um viés na expressão gênica, o que leva a interpretação errada dos resultados, uma vez que a expressão deste gene pode se apresentar de forma diferente no tecido normal. Conforme a representatividade tumoral (acima de 60-70%) aumenta, a análise atinge qualidade suficiente para a avaliação da expressão gênica e obtenção de dados fidedignos e reais de expressão82, 83. A fim de evitar interferência em nossa análise, atribuída a presença de tecido tumoral, optamos por incluir amostras com representatividade de tecido tumoral acima de 50%. De fato, podemos avaliar que a maioria das nossas amostras (71%) foram classificadas com mais de 80% de tecido tumoral. Dessa forma, podemos inferir que os dados de expressão dos genes avaliados em nosso estudo são reais e refletem o comportamento destes genes nos tumores, não tendo uma interferência significativa proveniente de células normais. Para a extração do RNA, utilizamos o método do TRIZOL de acordo com as orientações do fabricante em todas as etapas, conforme descrito na metodologia. O TRIZOL é um dos produtos comerciais mais utilizados para esse fim. Ele consiste de uma solução monofásica de fenol e guanidina isotiocianato que atua provocando a lise das células mantendo a integridade do RNA. A vantagem da utilização deste método é o maior rendimento em relação a quantidade de RNA resgatado. A principal desvantagem é a possibilidade de contaminação do RNA com DNA genômico, uma vez que a separação do RNA é realizada manualmente, independente do uso de coluna. Diversos trabalhos na literatura, utilizam o 54 TRIZOL para a extração de RNA de amostras tumorais para avaliar a expressão de genes por RT-qPCR, inclusive em osteossarcoma30, 33, 84-86. Ao avaliarmos a qualidade do RNA extraído por TRIZOL, nos deparamos com a maior parte das amostras apresentando o RNA de parcialmente a totalmente degradado. Com o intuito de obter um RNA de melhor qualidade, optamos por re-extrair algumas amostras por meio de um kit comercial de extração de RNA. O kit de extração é um método rápido de isolamento e purificação do RNA baseado no processo de colunas cromatográficas utilizando uma resina como matriz de separação. A vantagem deste método é que o RNA purificado apresenta alta pureza, mas a principal desvantagem é o menor rendimento em termos de quantidade de RNA. Mesmo com a utilização do kit de extração, nossos resultados mostraram que não houve melhora significativa na qualidade do RNA extraído (p=0,164). A integridade do RNA extraído, comumente é avaliada por meio de gel de agarose, porém, essa técnica é subjetiva e pouco reprodutível87. De maneira crescente na literatura, o padrão ouro para avaliar a integridade do RNA tem sido a utilização do Bioanalyzer, um equipamento capaz de separar pequenas quantidades de RNA de acordo com o seu peso molecular em canais presentes em um chip, para então serem analisadas por fluorescência. O resultado é dado através de uma curva de eletroesferograma e um algoritmo denominado RIN (RNA Integrity Number) que indica a qualidade do RNA87, 88. Para muitas técnicas utilizadas nos estudos de expressão gênica o RNA intacto é essencial, mas de acordo com alguns autores, recomenda-se um valor de RIN superior a 5 como requisito mínimo para uma quantificação por RT-qPCR de sucesso e de confiança87-89. Além disso, a utilização de iniciadores e sondas para a amplificação de fragmentos menores do que 100pb associados a análise de PCR em tempo real, no nosso caso, permitiu a utilização de amostras com RIN> 4, com resultados satisfatórios para as análises de expressão dos genes como demonstrado em nossos resultados. Uma vez que as amostras apresentam um certo grau de degradação, outro ponto importante é a quantificação do RNA, a qual inicialmente foi realizada por espectrofotômetro (NanoVue). Ao avaliarmos a expressão dos normalizadores, detectamos uma variação na expressão desses genes, com um desvio padrão muito alto. Isso nos levou a realizar a quantificação do RNA pelo método de fluorescência (Qubit), já que o espectrofotômetro é menos específico uma vez que qualquer partícula na solução poderá influenciar na leitura e fornecer concentrações superestimadas. 55 A partir dessa nova quantificação, notamos que a concentração de RNA fornecida pelo Qubit era menor do que a indicada pelo espectrofotômetro, explicando em parte, o alto desvio padrão encontrado na avaliação da expressão dos normalizadores. Sendo assim, concluímos ser a melhor opção a quantificação obtida pelo Qubit e dessa forma, observamos uma menor variação para um dos normalizadores (ACTB). Com essa estratégia podemos afirmar que a variação de expressão dos genes avaliados entre as amostras é real e não foi gerada a partir de artefatos (diferença na quantidade de RNA) devido a degradação das amostras. 6.2 Caracterização das amostras Nos últimos 25 anos, notou-se uma melhora significativa no prognóstico de pacientes com OS, principalmente naqueles com doença localizada. Com a associação da quimioterapia a cirurgia e com regimes de quimioterapia utilizando multidrogas, e doses mais elevadas, a sobrevida em 5 anos desses pacientes aumentou de forma progressiva até a década de 80. No entanto, estudos publicados após essa década, mostraram que não houve melhoras significativas na sobrevivência de pacientes com OS e destacam a importância de uma nova estratégia na gestão sistêmica dessa condição ainda muito letal90, 91. Pelo amplo espectro de apresentação e prognósticos distintos observados no tratamento do OS, diversos fatores epidemiológicos, clínico-patológicos e marcadores biológicos têm sido buscados para melhor predizer a evolução dos pacientes e estimar sua sobrevida10, 92-94. Entre os fatores reconhecidos com importância prognóstica destacam-se: tamanho do tumor94, localização e resposta à quimioterapia neodajuvante17, 95. Outros fatores prognósticos desfavoráveis já foram descritos em alguns estudos e incluem: presença de doença metastática ao diagnóstico17, 34, 94, 96, tumores volumosos97, idade inferior a 10 anos e sexo masculino10, 12, 93. O perfil dos pacientes descritos no nosso trabalho quanto aos fatores prognósticos, não difere dos padrões observados em outros estudos publicados, com exceção do sexo, pois na maioria dos estudos, o sexo masculino costuma ser o mais frequentemente afetado17, 98. Assim, dos 36 pacientes incluídos no estudo, a maior frequência do osteossarcoma foi no sexo feminino (55,6%), e segundo Mirabello et al.1 que avaliou a 56 incidência e a sobrevida dos pacientes com OS de 1973 a 2004, também encontrou a maior frequência no sexo feminino. Apesar de alguns autores considerarem o sexo masculino como um prognóstico desfavorável10, 12, 93, a sobrevida global e a sobrevida livre de eventos em 5 anos foi maior para o sexo masculino, mas nossa análise não demonstrou resultado significativo que nos permitisse corroborar estes achados. Em relação à idade ao diagnóstico, 29 (80,6%) possuíam idade igual ou superior a 10 anos. A média de idade foi de 15 anos, o que condiz com os dados da literatura. Apesar da média de idade ao diagnóstico ser muito variável na maioria dos estudos17, 95, 99, 100, os dados reafirmam que o maior pico de incidência desta doença é na segunda década de vida1, 101, 102. A idade como fator prognóstico na literatura é muito controversa. Pacientes com menos de 14 anos e mais de 40 anos, segundo dois grandes estudos retrospectivos, apresentaram sobrevidas inferiores12, 103. Entretanto, em um estudo retrospectivo, multicêntrico, com 2680 pacientes de 10 centros de pesquisa do mundo, demonstrou que a idade não foi um marcador prognóstico93. No Brasil, em um estudo com 225 pacientes, a idade não mostrou ser um fator prognóstico17. No nosso estudo, a idade não impactou a sobrevida dos pacientes. O osteossarcoma pode acometer qualquer osso do esqueleto axial ou apendicular do corpo humano, mas preferencialmente acomete as metáfises dos ossos longos, como fêmur distal, tíbia e úmero proximais. Neste trabalho, a grande maioria 91,7% (33/36) apresentou comprometimento de ossos longos, principalmente fêmur e tíbia, seguido do úmero, além do predomínio da histologia clássica, corroborando os dados da literatura17, 90, 95. De acordo com a literatura, tumores axiais possuem um pior prognóstico quando comparados com os de extremidades, principalmente pela dificuldade de ressecção total do tumor axial. Osteossarcoma de pelve, coluna e tórax tem uma sobrevida de apenas 20-40% quando comparado com o de extremidades (60-70%)12, 104, 105. Em nosso trabalho, não temos tumores localizados em região do esqueleto axial, não sendo possível realizar essa associação com a sobrevida dos pacientes. Entretanto, nossos dados sugerem que mesmo considerando apenas o esqueleto apendicular, a presença de tumor em região femural apresenta SG e SLE aproximadamente 30% menor em 5 anos, comparados aos tumores localizados em tíbia, fíbula e úmero. Historicamente, acreditava-se que os subtipos histológicos clássicos de osteossarcoma (osteoblástico, fibroblástico e condroblástico) apresentavam mesmo 57 comportamento. No entanto, há dados que sugerem que o osteossarcoma condroblástico esta associado a pior resposta à quimioterapia em comparação com outros subtipos e, portanto, está associado a um mau prognóstico106. Nossos resultados não encontraram significância estatística, mas podemos notar que os pacientes que apresentaram uma pior sobrevida global e livre de eventos estavam no grupo do condroblástico/fibroblástico/ telangectásico. Das oito amostras deste grupo, cinco são do subtipo condroblástico e desses 80% (4) tem resposta pobre a quimioterapia (grau de Huvos 1/2), o que parece corroborar os dados da literatura. Para o paciente restante, esse critério não se aplicou pois não foi submetido a cirurgia. No tratamento do osteossarcoma, o grau de Huvos refere-se ao grau de necrose tumoral em resposta à quimioterapia neo-adjuvante107. É considerado pela literatura como um fator prognóstico muito importante na sobrevida dos pacientes com osteossarcoma92, 93, 97. O grau de Huvos é avaliado em porcentagem na análise histopatológica da peça cirúrgica do paciente, após o início do tratamento. É uma avaliação direta e objetiva da eficácia do tratamento quimioterápico. No nosso estudo, em relação ao grau de resposta à quimioterapia encontramos que dos 36 casos, apenas 2 não foram avaliados, pois os pacientes não foram submetidos à cirurgia pós quimioterapia. Assim, para aqueles em que esse parâmetro estava disponível, 35,3% (12/34) dos casos foram classificados como grau 1 e 41,17% (14/34) grau 2, ou seja, ~ 76,5% dos casos apresentou resposta pobre à quimioterapia (índice de necrose tumoral menor ou igual a 90%). Do restante, 17,64% (6/34) apresentaram grau 3 e 5,88% (2/34) grau 4 e foram classificados com boa resposta à quimioterapia (necrose tumoral maior que 90%). Para análise estatística, o grau de Huvos foi dividido em duas categorias: boa resposta à quimioterapia (grau 3 e 4) com somente 22,5 % (8/34) dos casos e pobre resposta à quimioterapia (grau 1 e 2) com 76,4% (26/34) dos casos. De acordo com a literatura, pacientes que alcançam pobre resposta ao tratamento, apresentam uma pior sobrevida quando comparado aos pacientes com boa resposta17, 108. No nosso estudo, o grau de Huvos não foi associado a sobrevida dos pacientes, mas podemos observar que a sobrevida global e livre de eventos foi maior em pacientes que responderam melhor ao tratamento (grau 3/4). Em relação ao tamanho do tumor, alguns estudos demonstraram que ele pode se correlacionar com o prognóstico: tumores maiores foram associados com estimativas inferiores de sobrevida109, 110. Tumores maiores que 10 cm tem sido associados com resposta 58 pobre ao tratamento em alguns estudos111, 112. Outros associaram tamanhos maiores que 12cm17. Entretanto, existe uma dificuldade em se estabelecer um ponto de corte onde a partir de um tamanho, essa variável se torne importante para indicar o prognóstico dos pacientes. Isto se deve às diversas técnicas utilizadas na mensuração do tamanho do tumor e somado a isso, o uso de diferentes modalidades de imagem como a radiografia convencional, a tomografia computadorizada e a ressonância magnética 108. No nosso estudo, o ponto de corte selecionado foi 12 cm, de acordo com o GBTO 17 e o método utilizado para a mensuração do tamanho do tumor foi preferencialmente a ressonância quando disponível, seguido pela tomografia e radiografia, neste caso quando apenas este exame estava disponível. No nosso trabalho, o tamanho do tumor não foi um fator prognóstico e podemos observar que pacientes com tumores maiores que 12 cm apresentam uma melhor sobrevida global e livre de eventos. Apesar de ter sido avaliado pelo mesmo radiologista, esse achado pode ser explicado pelo fato de utilizarmos diferentes métodos/exames de imagem para determinar o tamanho de tumor ao diagnóstico. A radiografia convencional proporciona ao radiologista uma menor precisão para a mensuração do tamanho do tumor, o que pode ter subestimado o tamanho de tumor de alguns pacientes (9/28). Ainda, para 8 dos 36 pacientes não havia disponível qualquer exame de imagem para a determinação do tamanho do tumor, o que restringiu a nossa amostragem para a análise deste aspecto. A presença de doença metastática tem sido considerada como fator prognóstico, e menos de 20% dos pacientes têm sobrevida livre de doença em 5 anos 12. Em relação à sobrevida global e livre de eventos de pacientes metastáticos e não metastáticos ao diagnóstico, nossos dados são comparáveis aos dados da literatura1,17 e podemos ver claramente que a doença metastática ao diagnóstico confere ao paciente uma sobrevida significativamente menor quando comparado ao não metastático. 6.3 Expressão dos lncRNAs em osteossarcoma Devido a raridade da doença, a maioria dos trabalhos que tem por objetivo estudar alterações moleculares em osteossarcoma tem à disposição casuísticas restritas em torno de 20 a 50 pacientes30, 33, 85, 99, 111, 112. Em nosso caso, apesar da disponibilidade de 175 amostras de tecido tumoral congelado de osteossarcoma, optamos por ser estringentes nos critérios de inclusão destas amostras no estudo, principalmente no que diz respeito à qualidade do 59 RNA. Dessa forma, minimizamos a introdução de viés nas análises de expressão gênica, devido à baixa qualidade do RNA, garantindo que o resultado obtido é fidedigno e reflete a real expressão destes genes nas amostras avaliadas. Diversos trabalhos que avaliam a expressão gênica em OS não descrevem a utilização de tais critérios em suas análises63, 99, 113, 114. 6.3.1 Correlação da expressão do BCYRN1 com as características clínico-patológicas e sobrevida O BCYRN1 é um RNA não codificador seletivamente expresso no sistema nervoso central de primatas e foi identificado em domínios somatodendríticos de um subconjunto de neurônios80. Na literatura, estudos avaliando a expressão de BCYRN1 em tumores humanos são escassos. Um dos primeiros estudos apenas avaliou a expressão de BCYRN1 em 19 tipos diferentes de tumor e revelou sua maior expressão nos carcinomas de mama, colo do útero, esôfago, pulmão e ovário, mas não nos tecidos normais correspondentes80. Posteriormente, outro grupo avaliou a expressão de BCYRN1 em tecido mamário normal em relação a carcinomas ductais in situ de baixo grau, alto grau e carcinomas invasivos. Os resultados mostraram que o BCYRN1 não é expresso em mama normal e não tem expressão significativa em carcinomas ductais in situ de baixo grau, em contrapartida, está altamente expresso em carcinomas ductais in situ de alto grau e em todos os carcinomas invasivos. A alta expressão deste gene foi sugerida como marcador de invasividade, diagnóstico e prognóstico no câncer de mama. A superexpressão do BCYRN1 nestes tumores pode ser usada como indicador de progressão tumoral43. Portanto, apesar de poucos relatos, os primeiros estudos avaliando a expressão deste gene demonstram evidências de que sua maior expressão ocorre nos tecidos tumorais e, ainda que essa expressão está associada com a agressividade do tumor, visto que sua maior expressão foi observada nos subtipos de alto grau e invasivos. Nós não encontramos na literatura estudos que avaliaram a expressão de BCYRN1 em osteossarcoma. No nosso estudo, quando comparamos a média de expressão de BCYRN1 entre as características avaliadas apenas encontramos associação em relação a idade. Os tumores de pacientes com > a 10 anos tem maior expressão do gene comparado ao grupo menor do que 10 anos (p=0,036). Não houve associação estatisticamente significativa com 60 as outras características avaliadas (tabela 6). Em relação ao padrão de expressão do gene (análise categórica) nossos resultados demonstraram que alta expressão está associada com os subtipos condroblástico, fibroblástico ou telangectásico (p=0,046 – Tabela 12). Como já descrito anteriormente, a literatura aponta o subtipo condroblástico associado a resposta pobre ao tratamento e pior prognóstico. Apesar de não significativo, nossos dados apontam que pacientes com este subtipo tumoral apresentam estimativas de SG e SLE 30% menores quando comparados ao osteoblástico (SG: 33,3% vs 57,0% p=0,225 e SLE: 28,6 vs 59,2 p=0,1, respectivamente). Nossos dados podem ser as primeiras evidências de que a expressão deste gene pode estar associado a um pior prognóstico dos pacientes com OS. Apesar de não significativos, outros achados reforçam esta hipótese. Por exemplo, pacientes pouco responsivos ao tratamento (Huvos 1/2) apresentaram expressão em média três vezes maior de BCYRN1 em relação aos que tem boa resposta (Huvos 3/4). Além disso, pacientes metastáticos ao diagnóstico e com metástase durante o tratamento apresentaram em média duas vezes e quatro vezes, respectivamente, maior expressão de BCYRN1 quando comparados aos não metastáticos (Tabela 6). Os pacientes que apresentaram maior expressão do gene tiveram estimativas de SG e SLE 20% menor em 5 anos em relação aos que tiveram baixa expressão (SG: 40,4 vs 59,4 p=0,295 e SLE: 40,2 vs 61 p=0,313, respectivamente). Embora a maioria das análises não sejam significativas, esses dados em conjunto apontam para uma possível associação entre a maior expressão do BCYRN1 como um potencial marcador de pior prognóstico nos pacientes com osteossarcoma. Sendo assim, a expressão e função deste gene merece uma investigação mais profunda em OS. 6.3.2 Correlação da expressão do HOTAIR com as características clínico-patológicas e sobrevida Relatos sugerem que o HOTAIR atua na regulação epigenética pelo recrutamento de complexos remodeladores de cromatina (polycomb repressive complex 2) sobre sequências alvo específicas que culmina na supressão de diversos genes44. Recentemente, foi demonstrado que o aumento de expressão de HOTAIR, induzido por TGF-β1, regula a ativação do processo de transição epitélio-mesenquima (TEM) em linhagens celulares de carcinoma colorretal e de mama115. TEM é um importante processo que permite as células epiteliais desativarem as ligações intercelulares e adquirir propriedades mesenquimais como 61 capacidade migratória aumentada e potencial metastático116. Também foi demostrado que a ativação da via de sinalização de TGF-β promove a transição epitélio-mesenquima, aumento de mobilidade celular e invasão na linhagem celular de osteossarcoma U2OS117 e que, a inibição dessa via resulta no bloqueio do crescimento do tumor primário e o desenvolvimento de metástases pulmonares em OS118. Apesar de sua função ainda não ter sido explorado em OS, esses relatos podem indicar um potencial papel funcional do HOTAIR em OS. De fato, o aumento de expressão de HOTAIR tem sido associado a presença de metástases e pior prognóstico em diversos tipos tumorais64, 119-121. Por exemplo, um dos primeiros relatos a avaliar a expressão de HOTAIR em tumores humanos, demonstrou que a superexpressão de HOTAIR em tumores primários de mama está associado ao desenvolvimento de mestástase e menor sobrevida global64. Em tumores primários de pulmão não pequenas células a alta expressão de HOTAIR foi relacionada com uma sobrevida significativamente menor dos pacientes119. Em relação a sobrevida, o mesmo foi observado para carcinoma escamoso de esôfago, além da maior expressão de HOTAIR também estar associada a um maior tamanho do tumor, estadiamento clínico mais avançado, presença de metástase linfonodal e menor grau de diferenciação histológica120. Em tumores gástricos, também foi descrita associação entre alta expressão de HOTAIR com presença de metástase linfonodal e estágio avançado da doença121. Apesar de todos esses achados que evidenciam a expressão de HOTAIR como um marcador de metástase e pior prognóstico, interessantemente, um estudo recente em tumores de mama demonstrou que não houve correlação significativa entre a superexpressão do HOTAIR com as características clínico-patológicas avaliadas. Ainda, de forma surpreendente demonstrou na análise multivariada que pacientes com maior expressão do HOTAIR tiveram menor risco de recidiva e morte, quando comparados àqueles com baixa expressão. Sendo assim, os autores sugerem que a expressão do HOTAIR pode não ser um marcador independente de prognóstico, sendo necessários estudos independentes de validação em tumores de mama122. Em relação aos sarcomas, um trabalho avaliou a expressão de HOTAIR em tumores primários e em metástases. Os autores demonstraram que menores níveis do HOTAIR foram associados com boa resposta ao tratamento. Neste mesmo estudo, a alta expressão do 62 HOTAIR foi relacionada a uma maior probabilidade de presença de metástases 123. Em relação a avaliação da expressão deste gene em OS, nada foi encontrado. No nosso estudo, quando a expressão do HOTAIR foi considerada como uma variável numérica, não encontramos associação significativa entre a média de expressão de HOTAIR e as características epidemiológicas e clínico-patológicas avaliadas. Apesar disso, vale a pena destacar nesta análise que a expressão de HOTAIR foi em média 5,4 vezes maior em pacientes com resposta pobre ao tratamento (Huvos 1/2) comparado aos bons respondedores (Huvos 3/4) (p=0,256 – Tabela 7), o que parece estar em concordância com os achados em relação a expressão deste gene em sarcomas123. Em relação a análise categórica de expressão do HOTAIR, nossos dados demonstraram que dos pacientes que apresentavam alta expressão do gene, 75% tinham tumores inferiores a 12cm (p=0,049 – Tabela 13). Na literatura, esse dado é um pouco controverso, uma vez que alguns estudos descrevem que não há associação significativa entre alta expressão do HOTAIR e o tamanho do tumor64, 122, 123, enquanto outros estudos descrevem essa associação120, 124, 125. De qualquer forma, esse achado deve ser avaliado de forma cuidadosa, uma vez que para essa característica não foi possível resgatar a informação em oito pacientes e, ainda, do restante há uma variação nos exames utilizados para a determinação do tamanho tumoral, como descrito anteriormente. Em relação as análises de sobrevida, ainda que não significativo, a SG e SLE nos pacientes que apresentaram alta expressão do gene foi inferior quando comparado com aqueles que tiveram baixa expressão (SG: 40,6% vs 64,8% p=0,242; SLE: 41,3% vs 62,3% p=0,224, respectivamente). Mesmo que não significativos, nossos dados demonstraram que a maior expressão do HOTAIR pode ser observada em pacientes que respondem pior ao tratamento e que apresentam menores estimativas de sobrevida, também podendo ser um potencial biomarcador de prognóstico em OS. Da mesma forma, a expressão e função deste gene merece uma investigação mais abrangente em OS. 6.3.3 Correlação da expressão do HULC com as características clínico-patológicas e sobrevida Ao avaliarmos a expressão do HULC, não encontramos nenhuma associação significativa com as variáveis clínico-patológicas estudadas. Podemos notar que mesmo não obtendo resultados significativos, a maior média de expressão dos genes estava relacionada 63 com pacientes considerados bons respondedores (grau 3 e 4), com idade igual ou superior a 10 anos e com ausência de metástase ao diagnóstico. Quando avaliamos as mesmas variáveis após categorização da expressão, notamos que a maioria dos pacientes que apresentaram alta expressão do gene pertencia ao tipo histológico osteoblástico, com idade igual ou superior a 10 anos (p=0,088) e com ausência de metástase ao diagnóstico (p=0,180). De acordo com a literatura, no câncer pancreático a alta expressão do HULC foi associada ao tamanho do tumor, metástase linfonodal e invasão vascular 79. No câncer gástrico, a alta expressão deste gene também foi relacionada com a presença de metástase linfonodal, metástase à distância e ao estadio mais avançado da doença. Foi observado os efeitos da superexpressão e do “knockdown” da expressão do HULC e determinou-se que este gene desempenha um papel importante na proliferação, migração, invasão e na inibição da apoptose. Neste estudo não foi encontrada associação do gene com outros fatores clínico-patológicos como idade, sexo, tamanho do tumor, localização e diferenciação do tumor78. No carcinoma hepatocelular, a expressão tecidual e plasmática de HULC estava aumentada entre pacientes com hepatocarcinoma e um grupo controle. Além de serem constatados níveis significativamente maiores de HULC no tecido/plasma de pacientes com hepatocarcinoma, houve relação direta entre os valores de expressão do HULC e outras variáveis, como o grau de diferenciação histológica. Estes resultados sugerem um potencial uso deste RNA como biomarcador diagnóstico e/ou prognóstico. Além disso, sua expressão foi maior quando comparada com tecidos normais do fígado e mostrou que a expressão deste gene pode estar associada com a agressividade e a progressão do tumor77. Na literatura, não encontramos relatos sobre a expressão do HULC em amostras de osteossarcoma. No nosso trabalho, não encontramos nenhum correlação significativa entre a expressão deste gene e um pior prognóstico, como sugere a literatura. 6.3.4 Correlação da expressão do MALAT1 com as características clínico-patológicas e sobrevida MALAT1 foi o primeiro lncRNA associado com um alto potencial metastático e pior prognóstico em um estudo avaliando pacientes portadores de câncer de pulmão de não pequenas células com ou sem metástase. MALAT1 mostrou estar superexpresso em muitos 64 tumores sólidos, dentre eles, mama, próstata, cólon, fígado, útero, hepatocarcinoma, pâncreas e osteossarcoma57, 58, 126-128. No nosso estudo, a maior expressão do MALAT1 foi associada significativamente a pacientes com idade igual ou superior a 10 anos quando analisamos a média da expressão (p=0,048), e após a categorização da expressão (p=0,008). Na literatura, não encontramos associação significativa deste gene com a idade63, 129-132. Apesar de não encontrarmos associação significativa com as demais características clínico-patológicas, observamos uma maior expressão do MALAT1 em pacientes do sexo masculino, em tumores localizados na fíbula, tíbia ou úmero. MALAT1 se mostrou com uma expressão um pouco aumentada em pacientes não metastáticos quando avaliamos a presença de metástase independente do momento diagnosticado. Entretanto, após a estratificação dos pacientes metastáticos, pudemos observar que a maior expressão do gene ocorre nos tumores de pacientes que desenvolvem metástase durante o tratamento. Além disso, observamos também que a maioria dos pacientes com baixa expressão não obtiveram boa resposta à quimioterapia (grau 1/2). O mecanismo de ação molecular do MALAT1 é ainda muito discutido. Em linhagem de células tumorais humanas, a inibição de MALAT1 suprimiu a migração e invasão57. Ao injetar em camundongos, xenoenxertos de células tumorais de câncer de pulmão não pequenas células, a formação e o crescimento dos tumores se mostraram prejudicada com a diminuição da expressão do MALAT1130. Diversos estudos têm mostrado uma forte associação da superexpressão do MALAT1 com a presença de metástase e um prognóstico ruim57, 63, 99, 129-132. Um estudo pioneiro com MALAT1 propôs identificar diferenças na expressão gênica entre tumores de pulmão não pequenas células que metastatizaram ou não e encontrou uma expressão maior (cerca de três vezes mais) nas amostras de pacientes que desenvolveram metástases, além da drástica piora na sobrevida global57. A superexpressão de MALAT1 foi avaliada em amostras de tumores como mieloma múltiplo e revelou que este lncRNA possui um papel na patogênese da doença e que a alteração na expressão desse gene depois do tratamento foi clinicamente significativa e pode servir como um preditor molecular dos pacientes em risco de progressão precoce. Além disso, este estudo revelou que pacientes com menor expressão do MALAT1 tinham 65 significativamente maior sobrevida livre de progressão em comparação com os pacientes com uma menor alteração na expressão62. Em osteossarcoma, um estudo que teve como objetivo demonstrar a associação entre os níveis de expressão de MALAT1 e características clínico-patológicas encontrou que a expressão do gene nos tecidos tumorais foi significativamente maior do que em tecidos normais; a presença de metástases pulmonares e acometimento linfonodal foram significativamente mais comuns no grupo de maior expressão; não houve associação entre a expressão do gene e o sexo, tamanho do tumor e sobrevida em 5 anos, e em relação a idade, o grupo com maior expressão do MALAT1 foi o mais jovem (em média, 16 anos) (p=0,05). Neste estudo, o autor concluiu que o osteossarcoma apresenta expressão elevada de MALAT1 e que o nível de expressão associa-se a uma maior probabilidade de desenvolver metástases pulmonares e acometimento linfonodal63. Outro estudo em osteossarcoma mostrou que a expressão do MALAT1 foi significativamente maior no grupo de maus respondedores ao tratamento quimioterápico, mas não encontrou associação com a sobrevida global e livre de eventos, assim como no nosso estudo99. 6.4 Limitações do estudo Como podemos observar, para a maioria das análises desenvolvidas em nosso estudo não foi possível encontrar associações significativas indicando o papel prognóstico destes genes em OS. Entretanto, uma limitação do nosso projeto foi o pequeno número de amostras (36) que pôde ser avaliado, uma vez que temos no biobanco da instituição amostras tumorais congeladas provenientes de 175 pacientes (os motivos da exclusão dessas amostras já foi descrito anteriormente). Uma parte considerável dessas amostras foi excluído devido aos restringentes critérios de qualidade determinados para que as amostras fossem utilizadas em nossas análises. Em contrapartida, esses critérios nos permitem afirmar com segurança, que nossos dados refletem com fidelidade a expressão destes genes em OS. Somado a essa pequena amostragem, temos o fato de que 35% dos pacientes apresentam um tempo de seguimento menor do que dois anos, o que pode não ser tempo suficiente para um paciente acometido por OS apresentar metástase ou óbito. Reconhecemos tais limitações e sugerimos que o aumento da amostragem, principalmente com a inclusão de pacientes com um tempo maior de seguimento pode nos revelar o real papel prognóstico destes genes em OS. 66 7 CONCLUSÕES Não houve diferença em termos de qualidade na extração do RNA entre o TRIZOL e método comercial baseado em coluna; A quantificação das amostras utilizando o Qubit é mais precisa e deve ser utilizada nas análises de expressão gênica quando o RNA apresenta degradação parcial; A maior expressão de BCYRN1 foi associada significativamente a pacientes com idade acima de 10 anos e com o subtipo condroblástico/fibroblástico/telangectásico; A expressão de HOTAIR não tem associação significativa com as características clínico- patológicas avaliadas; A expressão de HULC não tem associação significativa com as características clínico- patológicas avaliadas; A maior expressão de MALAT1 foi associada significativamente a pacientes com idade acima de 10 anos; As expressões de BCYRN1, HOTAIR, HULC e MALAT1 não foram significativamente associadas a SG e SLE dos pacientes. 67 REFERÊNCIAS BIBLIOGRÁFICAS 1. Mirabello L, Troisi RJ, Savage SA. Osteosarcoma incidence and survival rates from 1973 to 2004: data from the Surveillance, Epidemiology, and End Results Program. Cancer. 2009;115(7):1531-43. 2. Stiller CA, Bielack SS, Jundt G, Steliarova-Foucher E. Bone tumours in European children and adolescents, 1978-1997. Report from the Automated Childhood Cancer Information System project. Eur J Cancer. 2006;42(13):2124-35. 3. Janeway KA, Barkauskas DA, Krailo MD, Meyers PA, Schwartz CL, Ebb DH, et al. Outcome for adolescent and young adult patients with osteosarcoma: a report from the Children's Oncology Group. Cancer. 2012;118(18):4597-605. 4. INCA. Coordenação de Prevenção e Vigilância de Câncer - Câncer da criança e adolescente no Brasil: dados dos registros de base populacional e de mortalidade. Rio de Janeiro, Brasil: Instituto Nacional de Câncer (INCA); 2008. 5. FOSP. Funadação Oncocentro de São Paulo - Registro Hospitalar de Câncer Pediátrico. São Paulo, Brasil2012. 6. FLETCHER CDM, UNNI KK. Pathology and genetics of tumours of soft tissue and bone. In: MERTENS F, editor. World Health Organization Classification of Tumours. Lyon, France: IARC Press; 2002. 7. VIGORITA VJ. Orthopaedic Pathology. Philadelphia, USA: Lippincott, Williams & Wilkings; 2008. 8. Klein MJ, Siegal GP. Osteosarcoma: anatomic and histologic variants. Am J Clin Pathol. 2006;125(4):555-81. 9. Friedman MA, Carter SK. The therapy of osteogenic sarcoma: current status and thoughts for the future. J Surg Oncol. 1972;4(5):482-510. 10. Petrilli AS, Gentil FC, Epelman S, Lopes LF, Bianchi A, Lopes A, et al. Increased survival, limb preservation, and prognostic factors for osteosarcoma. Cancer. 1991;68(4):733-7. 11. Meyers PA, Gorlick R. Osteosarcoma. Pediatr Clin North Am. 1997;44(4):973-89. 12. Bielack SS, Kempf-Bielack B, Delling G, Exner GU, Flege S, Helmke K, et al. Prognostic factors in high-grade osteosarcoma of the extremities or trunk: an analysis of 1,702 patients treated on neoadjuvant cooperative osteosarcoma study group protocols. J Clin Oncol. 2002;20(3):776-90. 13. Harting MT, Blakely ML. Management of osteosarcoma pulmonary metastases. Semin Pediatr Surg. 2006;15(1):25-9. 68 14. Hughes DP. Strategies for the targeted delivery of therapeutics for osteosarcoma. Expert Opin Drug Deliv. 2009;6(12):1311-21. 15. Bacci G, Rocca M, Salone M, Balladelli A, Ferrari S, Palmerini E, et al. High grade osteosarcoma of the extremities with lung metastases at presentation: treatment with neoadjuvant chemotherapy and simultaneous resection of primary and metastatic lesions. J Surg Oncol. 2008;98(6):415-20. 16. Kager L, Zoubek A, Pötschger U, Kastner U, Flege S, Kempf-Bielack B, et al. Primary metastatic osteosarcoma: presentation and outcome of patients treated on neoadjuvant Cooperative Osteosarcoma Study Group protocols. J Clin Oncol. 2003;21(10):2011-8. 17. Petrilli AS, de Camargo B, Filho VO, Bruniera P, Brunetto AL, Jesus-Garcia R, et al. Results of the Brazilian Osteosarcoma Treatment Group Studies III and IV: prognostic factors and impact on survival. J Clin Oncol. 2006;24(7):1161-8. 18. Briccoli A, Rocca M, Salone M, Guzzardella GA, Balladelli A, Bacci G. High grade osteosarcoma of the extremities metastatic to the lung: long-term results in 323 patients treated combining surgery and chemotherapy, 1985-2005. Surg Oncol. 2010;19(4):193-9. 19. Bielack SS, Kempf-Bielack B, Branscheid D, Carrle D, Friedel G, Helmke K, et al. Second and subsequent recurrences of osteosarcoma: presentation, treatment, and outcomes of 249 consecutive cooperative osteosarcoma study group patients. J Clin Oncol. 2009;27(4):557-65. 20. Martin JW, Squire JA, Zielenska M. The genetics of osteosarcoma. Sarcoma. 2012;2012:627254. 21. Marina N, Gebhardt M, Teot L, Gorlick R. Biology and therapeutic advances for pediatric osteosarcoma. Oncologist. 2004;9(4):422-41. 22. Jacks T, Remington L, Williams BO, Schmitt EM, Halachmi S, Bronson RT, et al. Tumor spectrum analysis in p53-mutant mice. Curr Biol. 1994;4(1):1-7. 23. Olive KP, Tuveson DA, Ruhe ZC, Yin B, Willis NA, Bronson RT, et al. Mutant p53 gain of function in two mouse models of Li-Fraumeni syndrome. Cell. 2004;119(6):847-60. 24. Eischen CM, Rehg JE, Korsmeyer SJ, Cleveland JL. Loss of Bax alters tumor spectrum and tumor numbers in ARF-deficient mice. Cancer Res. 2002;62(7):2184-91. 25. Chen W, Cooper TK, Zahnow CA, Overholtzer M, Zhao Z, Ladanyi M, et al. Epigenetic and genetic loss of Hic1 function accentuates the role of p53 in tumorigenesis. Cancer Cell. 2004;6(4):387-98. 26. Wong FL, Boice JD, Abramson DH, Tarone RE, Kleinerman RA, Stovall M, et al. Cancer incidence after retinoblastoma. Radiation dose and sarcoma risk. JAMA. 1997;278(15):12627. 69 27. Siddiqui R, Onel K, Facio F, Nafa K, Diaz LR, Kauff N, et al. The TP53 mutational spectrum and frequency of CHEK2*1100delC in Li-Fraumeni-like kindreds. Fam Cancer. 2005;4(2):177-81. 28. Paulino AC, Fowler BZ. Secondary neoplasms after radiotherapy for a childhood solid tumor. Pediatr Hematol Oncol. 2005;22(2):89-101. 29. Broadhead ML, Clark JC, Myers DE, Dass CR, Choong PF. The molecular pathogenesis of osteosarcoma: a review. Sarcoma. 2011;2011:959248. 30. Tesser-Gamba F, Petrilli AS, de Seixas Alves MT, Filho RJ, Juliano Y, Toledo SR. MAPK7 and MAP2K4 as prognostic markers in osteosarcoma. Hum Pathol. 2012;43(7):994-1002. 31. Egas-Bejar D, Anderson PM, Agarwal R, Corrales-Medina F, Devarajan E, Huh WW, et al. Theranostic Profiling for Actionable Aberrations in Advanced High Risk Osteosarcoma with Aggressive Biology Reveals High Molecular Diversity: The Human Fingerprint Hypothesis. Oncoscience. 2014;1(2):167-79. 32. Huether R, Dong L, Chen X, Wu G, Parker M, Wei L, et al. The landscape of somatic mutations in epigenetic regulators across 1,000 paediatric cancer genomes. Nat Commun. 2014;5:3630. 33. Salinas-Souza C, De Oliveira R, Alves MT, Garcia Filho RJ, Petrilli AS, Toledo SR. The metastatic behavior of osteosarcoma by gene expression and cytogenetic analyses. Hum Pathol. 2013;44(10):2188-98. 34. Bridge JA, Nelson M, McComb E, McGuire MH, Rosenthal H, Vergara G, et al. Cytogenetic findings in 73 osteosarcoma specimens and a review of the literature. Cancer Genet Cytogenet. 1997;95(1):74-87. 35. Squire JA, Pei J, Marrano P, Beheshti B, Bayani J, Lim G, et al. High-resolution mapping of amplifications and deletions in pediatric osteosarcoma by use of CGH analysis of cDNA microarrays. Genes Chromosomes Cancer. 2003;38(3):215-25. 36. BOEHM AK, NEFF JR, SQUIRE JA, BAYANI J, NELSON M, BRIDGE JA. Cytogenetic Findings in 36 Osteosarcoma Specimens and a Review of the Literature. Fetal and Pediatric Pathology. 2000;19(5):359-76. 37. Batanian JR, Cavalli LR, Aldosari NM, Ma E, Sotelo-Avila C, Ramos MB, et al. Evaluation of paediatric osteosarcomas by classic cytogenetic and CGH analyses. Mol Pathol. 2002;55(6):389-93. 38. Bertone P, Stolc V, Royce TE, Rozowsky JS, Urban AE, Zhu X, et al. Global identification of human transcribed sequences with genome tiling arrays. Science. 2004;306(5705):2242-6. 70 39. Kapranov P, Cheng J, Dike S, Nix DA, Duttagupta R, Willingham AT, et al. RNA maps reveal new RNA classes and a possible function for pervasive transcription. Science. 2007;316(5830):1484-8. 40. Calin GA, Croce CM. MicroRNA signatures in human cancers. Nat Rev Cancer. 2006;6(11):857-66. 41. Chen LL, Carmichael GG. Long noncoding RNAs in mammalian cells: what, where, and why? Wiley Interdiscip Rev RNA. 2010;1(1):2-21. 42. Kapranov P, Drenkow J, Cheng J, Long J, Helt G, Dike S, et al. Examples of the complex architecture of the human transcriptome revealed by RACE and high-density tiling arrays. Genome Res. 2005;15(7):987-97. 43. Iacoangeli A, Lin Y, Morley EJ, Muslimov IA, Bianchi R, Reilly J, et al. BC200 RNA in invasive and preinvasive breast cancer. Carcinogenesis. 2004;25(11):2125-33. 44. Rinn JL, Kertesz M, Wang JK, Squazzo SL, Xu X, Brugmann SA, et al. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell. 2007;129(7):1311-23. 45. Willingham AT, Orth AP, Batalov S, Peters EC, Wen BG, Aza-Blanc P, et al. A strategy for probing the function of noncoding RNAs finds a repressor of NFAT. Science. 2005;309(5740):1570-3. 46. Clemson CM, Hutchinson JN, Sara SA, Ensminger AW, Fox AH, Chess A, et al. An architectural role for a nuclear noncoding RNA: NEAT1 RNA is essential for the structure of paraspeckles. Mol Cell. 2009;33(6):717-26. 47. Gutschner T, Diederichs S. The hallmarks of cancer: a long non-coding RNA point of view. RNA Biol. 2012;9(6):703-19. 48. Jia H, Osak M, Bogu GK, Stanton LW, Johnson R, Lipovich L. Genome-wide computational identification and manual annotation of human long noncoding RNA genes. RNA. 2010;16(8):1478-87. 49. Lipovich L, Johnson R, Lin CY. MacroRNA underdogs in a microRNA world: evolutionary, regulatory, and biomedical significance of mammalian long non-protein-coding RNA. Biochim Biophys Acta. 2010;1799(9):597-615. 50. Fu X, Ravindranath L, Tran N, Petrovics G, Srivastava S. Regulation of apoptosis by a prostate-specific and prostate cancer-associated noncoding gene, PCGEM1. DNA Cell Biol. 2006;25(3):135-41. 51. Amaral PP, Neyt C, Wilkins SJ, Askarian-Amiri ME, Sunkin SM, Perkins AC, et al. Complex architecture and regulated expression of the Sox2ot locus during vertebrate development. RNA. 2009;15(11):2013-27. 71 52. Fidler IJ. The pathogenesis of cancer metastasis: the 'seed and soil' hypothesis revisited. Nat Rev Cancer. 2003;3(6):453-8. 53. Tripathi V, Ellis JD, Shen Z, Song DY, Pan Q, Watt AT, et al. The nuclear-retained noncoding RNA MALAT1 regulates alternative splicing by modulating SR splicing factor phosphorylation. Mol Cell. 2010;39(6):925-38. 54. Wilusz JE, Freier SM, Spector DL. 3' end processing of a long nuclear-retained noncoding RNA yields a tRNA-like cytoplasmic RNA. Cell. 2008;135(5):919-32. 55. Kuiper RP, Schepens M, Thijssen J, van Asseldonk M, van den Berg E, Bridge J, et al. Upregulation of the transcription factor TFEB in t(6;11)(p21;q13)-positive renal cell carcinomas due to promoter substitution. Hum Mol Genet. 2003;12(14):1661-9. 56. Hutchinson JN, Ensminger AW, Clemson CM, Lynch CR, Lawrence JB, Chess A. A screen for nuclear transcripts identifies two linked noncoding RNAs associated with SC35 splicing domains. BMC Genomics. 2007;8:39. 57. Ji P, Diederichs S, Wang W, Böing S, Metzger R, Schneider PM, et al. MALAT-1, a novel noncoding RNA, and thymosin beta4 predict metastasis and survival in early-stage non-small cell lung cancer. Oncogene. 2003;22(39):8031-41. 58. Lin R, Maeda S, Liu C, Karin M, Edgington TS. A large noncoding RNA is a marker for murine hepatocellular carcinomas and a spectrum of human carcinomas. Oncogene. 2007;26(6):851-8. 59. Gutschner T, Hämmerle M, Diederichs S. MALAT1 -- a paradigm for long noncoding RNA function in cancer. J Mol Med (Berl). 2013;91(7):791-801. 60. Tano K, Mizuno R, Okada T, Rakwal R, Shibato J, Masuo Y, et al. MALAT-1 enhances cell motility of lung adenocarcinoma cells by influencing the expression of motility-related genes. FEBS Lett. 2010;584(22):4575-80. 61. Guo F, Li Y, Liu Y, Wang J, Li G. Inhibition of metastasis-associated lung adenocarcinoma transcript 1 in CaSki human cervical cancer cells suppresses cell proliferation and invasion. Acta Biochim Biophys Sin (Shanghai). 2010;42(3):224-9. 62. Cho SF, Chang YC, Chang CS, Lin SF, Liu YC, Hsiao HH, et al. MALAT1 long non-coding RNA is overexpressed in multiple myeloma and may serve as a marker to predict disease progression. BMC Cancer. 2014;14:809. 63. Dong Y, Liang G, Yuan B, Yang C, Gao R, Zhou X. MALAT1 promotes the proliferation and metastasis of osteosarcoma cells by activating the PI3K/Akt pathway. Tumour Biol. 2014. 72 64. Gupta RA, Shah N, Wang KC, Kim J, Horlings HM, Wong DJ, et al. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature. 2010;464(7291):1071-6. 65. Geng YJ, Xie SL, Li Q, Ma J, Wang GY. Large intervening non-coding RNA HOTAIR is associated with hepatocellular carcinoma progression. J Int Med Res. 2011;39(6):2119-28. 66. Yang Z, Zhou L, Wu LM, Lai MC, Xie HY, Zhang F, et al. Overexpression of long noncoding RNA HOTAIR predicts tumor recurrence in hepatocellular carcinoma patients following liver transplantation. Ann Surg Oncol. 2011;18(5):1243-50. 67. Lee NK, Lee JH, Park CH, Yu D, Lee YC, Cheong JH, et al. Long non-coding RNA HOTAIR promotes carcinogenesis and invasion of gastric adenocarcinoma. Biochem Biophys Res Commun. 2014;451(2):171-8. 68. Zhou X, Chen J, Tang W. The molecular mechanism of HOTAIR in tumorigenesis, metastasis, and drug resistance. Acta Biochim Biophys Sin (Shanghai). 2014;46(12):1011-5. 69. He X, Bao W, Li X, Chen Z, Che Q, Wang H, et al. The long non-coding RNA HOTAIR is upregulated in endometrial carcinoma and correlates with poor prognosis. Int J Mol Med. 2014;33(2):325-32. 70. Kim HJ, Lee DW, Yim GW, Nam EJ, Kim S, Kim SW, et al. Long non-coding RNA HOTAIR is associated with human cervical cancer progression. Int J Oncol. 2015;46(2):521-30. 71. Xue Y, Gu D, Ma G, Zhu L, Hua Q, Chu H, et al. Genetic variants in lncRNA HOTAIR are associated with risk of colorectal cancer. Mutagenesis. 2014. 72. Zhang S, Chen S, Yang G, Gu F, Li M, Zhong B, et al. Long noncoding RNA HOTAIR as an independent prognostic marker in cancer: a meta-analysis. PLoS One. 2014;9(8):e105538. 73. Deng Q, Sun H, He B, Pan Y, Gao T, Chen J, et al. Prognostic value of long non-coding RNA HOTAIR in various cancers. PLoS One. 2014;9(10):e110059. 74. Panzitt K, Tschernatsch MM, Guelly C, Moustafa T, Stradner M, Strohmaier HM, et al. Characterization of HULC, a novel gene with striking up-regulation in hepatocellular carcinoma, as noncoding RNA. Gastroenterology. 2007;132(1):330-42. 75. Matouk IJ, Abbasi I, Hochberg A, Galun E, Dweik H, Akkawi M. Highly upregulated in liver cancer noncoding RNA is overexpressed in hepatic colorectal metastasis. Eur J Gastroenterol Hepatol. 2009;21(6):688-92. 76. Wang J, Liu X, Wu H, Ni P, Gu Z, Qiao Y, et al. CREB up-regulates long non-coding RNA, HULC expression through interaction with microRNA-372 in liver cancer. Nucleic Acids Res. 2010;38(16):5366-83. 73 77. Xie H, Ma H, Zhou D. Plasma HULC as a promising novel biomarker for the detection of hepatocellular carcinoma. Biomed Res Int. 2013;2013:136106. 78. Zhao Y, Guo Q, Chen J, Hu J, Wang S, Sun Y. Role of long non-coding RNA HULC in cell proliferation, apoptosis and tumor metastasis of gastric cancer: a clinical and in vitro investigation. Oncol Rep. 2014;31(1):358-64. 79. Peng W, Gao W, Feng J. Long noncoding RNA HULC is a novel biomarker of poor prognosis in patients with pancreatic cancer. Med Oncol. 2014;31(12):346. 80. Chen W, Böcker W, Brosius J, Tiedge H. Expression of neural BC200 RNA in human tumours. J Pathol. 1997;183(3):345-51. 81. Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 2001;25(4):402-8. 82. Yoshihara K, Shahmoradgoli M, Martínez E, Vegesna R, Kim H, Torres-Garcia W, et al. Inferring tumour purity and stromal and immune cell admixture from expression data. Nat Commun. 2013;4:2612. 83. Elloumi F, Hu Z, Li Y, Parker JS, Gulley ML, Amos KD, et al. Systematic bias in genomic classification due to contaminating non-neoplastic tissue in breast tumor samples. BMC Med Genomics. 2011;4:54. 84. Yang J, Annala M, Ji P, Wang G, Zheng H, Codgell D, et al. Recurrent LRP1-SNRNP25 and KCNMB4-CCND3 fusion genes promote tumor cell motility in human osteosarcoma. J Hematol Oncol. 2014;7(1):76. 85. Zhang Q, Geng PL, Yin P, Wang XL, Jia JP, Yao J. Down-regulation of long non-coding RNA TUG1 inhibits osteosarcoma cell proliferation and promotes apoptosis. Asian Pac J Cancer Prev. 2013;14(4):2311-5. 86. Toledo SR, Oliveira ID, Okamoto OK, Zago MA, de Seixas Alves MT, Filho RJ, et al. Bone deposition, bone resorption, and osteosarcoma. J Orthop Res. 2010;28(9):1142-8. 87. Mueller O, Hahnenberger K, Dittmann M, Yee H, Dubrow R, Nagle R, et al. A microfluidic system for high-speed reproducible DNA sizing and quantitation. Electrophoresis. 2000;21(1):128-34. 88. Opitz L, Salinas-Riester G, Grade M, Jung K, Jo P, Emons G, et al. Impact of RNA degradation on gene expression profiling. BMC Med Genomics. 2010;3:36. 89. Schroeder A, Mueller O, Stocker S, Salowsky R, Leiber M, Gassmann M, et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Mol Biol. 2006;7:3. 74 90. M L, F E. Osteosarcoma. In: P P, D P, editors. Principles and Practice of Pediatric Oncology. 4th ed ed. Philadelphia: Lippincott-Raven Publishers; 2002. p. 1051-89. 91. Allison DC, Carney SC, Ahlmann ER, Hendifar A, Chawla S, Fedenko A, et al. A metaanalysis of osteosarcoma outcomes in the modern medical era. Sarcoma. 2012;2012:704872. 92. Ferrari S, Bertoni F, Mercuri M, Picci P, Giacomini S, Longhi A, et al. Predictive factors of disease-free survival for non-metastatic osteosarcoma of the extremity: an analysis of 300 patients treated at the Rizzoli Institute. Ann Oncol. 2001;12(8):1145-50. 93. Pakos EE, Nearchou AD, Grimer RJ, Koumoullis HD, Abudu A, Bramer JA, et al. Prognostic factors and outcomes for osteosarcoma: an international collaboration. Eur J Cancer. 2009;45(13):2367-75. 94. FRS J, LS L, JAS L, MB R. Avaliação dos fatores prognósticos e sobrevida de pacientes com osteossarcoma atendidos em um Hospital Filantrópico de Teresina (PI) Brasil Rev Bras Ortop 2013;48 (1):87-91. 95. A R, CG CJ, J M, L G, L DL, A D, et al. Clinical features in osteosarcoma and prognostic implications. Journal of Pediatrics. 2004;80(1):65-70. 96. Meyer WH, Pratt CB, Poquette CA, Rao BN, Parham DM, Marina NM, et al. Carboplatin/ifosfamide window therapy for osteosarcoma: results of the St Jude Children's Research Hospital OS-91 trial. J Clin Oncol. 2001;19(1):171-82. 97. Bacci G, Mercuri M, Briccoli A, Ferrari S, Bertoni F, Donati D, et al. Osteogenic sarcoma of the extremity with detectable lung metastases at presentation. Results of treatment of 23 patients with chemotherapy followed by simultaneous resection of primary and metastatic lesions. Cancer. 1997;79(2):245-54. 98. ITJ F, TO B, GJA O, L C-M, ICJ G, PV M, et al. Factores de riesgo para los tumores óseos malignos pediátricos. Anales de Pediatría. 2005;63(6):537-47. 99. Fellenberg J, Bernd L, Delling G, Witte D, Zahlten-Hinguranage A. Prognostic significance of drug-regulated genes in high-grade osteosarcoma. Mod Pathol. 2007;20(10):1085-94. 100. CT F, AS P, CG D, MGR G. Itinerário terapêutico de adolescentes e adultos jovens com osteossarcoma. Revista Brasileira de Cancerologia. 2012;58(2):197-208. 101. Wu PK, Chen WM, Chen CF, Lee OK, Haung CK, Chen TH. Primary osteogenic sarcoma with pulmonary metastasis: clinical results and prognostic factors in 91 patients. Jpn J Clin Oncol. 2009;39(8):514-22. 102. OP C. Tumores osseos e lesões pseudotumorais. In: Hebert S XR, Pardini Junior AG, Barros Filho TEP, editor. Ortopedia e traumatologia: princípios e prática. 3a. edição ed. Porto Alegre: Artmed; 2003. p. 771-81. 75 103. Bacci G, Longhi A, Versari M, Mercuri M, Briccoli A, Picci P. Prognostic factors for osteosarcoma of the extremity treated with neoadjuvant chemotherapy: 15-year experience in 789 patients treated at a single institution. Cancer. 2006;106(5):1154-61. 104. Ozaki T, Flege S, Liljenqvist U, Hillmann A, Delling G, Salzer-Kuntschik M, et al. Osteosarcoma of the spine: experience of the Cooperative Osteosarcoma Study Group. Cancer. 2002;94(4):1069-77. 105. Ozaki T, Flege S, Kevric M, Lindner N, Maas R, Delling G, et al. Osteosarcoma of the pelvis: experience of the Cooperative Osteosarcoma Study Group. J Clin Oncol. 2003;21(2):334-41. 106. Bacci G, Bertoni F, Longhi A, Ferrari S, Forni C, Biagini R, et al. Neoadjuvant chemotherapy for high-grade central osteosarcoma of the extremity. Histologic response to preoperative chemotherapy correlates with histologic subtype of the tumor. Cancer. 2003;97(12):3068-75. 107. Huvos AG, Rosen G, Marcove RC. Primary osteogenic sarcoma: pathologic aspects in 20 patients after treatment with chemotherapy en bloc resection, and prosthetic bone replacement. Arch Pathol Lab Med. 1977;101(1):14-8. 108. Bakhshi S, Radhakrishnan V. Prognostic markers in osteosarcoma. Expert Rev Anticancer Ther. 2010;10(2):271-87. 109. Munajat I, Zulmi W, Norazman MZ, Wan Faisham WI. Tumour volume and lung metastasis in patients with osteosarcoma. J Orthop Surg (Hong Kong). 2008;16(2):182-5. 110. Lee JA, Kim MS, Kim DH, Lim JS, Yoo JY, Koh JS, et al. Relative tumor burden predicts metastasis-free survival in pediatric osteosarcoma. Pediatr Blood Cancer. 2008;50(2):195200. 111. Bramer JA, van Linge JH, Grimer RJ, Scholten RJ. Prognostic factors in localized extremity osteosarcoma: a systematic review. Eur J Surg Oncol. 2009;35(10):1030-6. 112. Bieling P, Rehan N, Winkler P, Helmke K, Maas R, Fuchs N, et al. Tumor size and prognosis in aggressively treated osteosarcoma. J Clin Oncol. 1996;14(3):848-58. 113. Li C, Zhan C, Chen Y, Fu Q, Zhu XD, He DW, et al. Analysis report for osteosarcoma expression profile. Eur Rev Med Pharmacol Sci. 2013;17(20):2804-9. 114. Namløs HM, Kresse SH, Müller CR, Henriksen J, Holdhus R, Sæter G, et al. Global gene expression profiling of human osteosarcomas reveals metastasis-associated chemokine pattern. Sarcoma. 2012;2012:639038. 115. Pádua Alves C, Fonseca AS, Muys BR, de Barros E Lima Bueno R, Bürger MC, de Souza JE, et al. Brief report: The lincRNA Hotair is required for epithelial-to-mesenchymal transition and stemness maintenance of cancer cell lines. Stem Cells. 2013;31(12):2827-32. 76 116. Celià-Terrassa T, Meca-Cortés O, Mateo F, de Paz AM, Rubio N, Arnal-Estapé A, et al. Epithelial-mesenchymal transition can suppress major attributes of human epithelial tumorinitiating cells. J Clin Invest. 2012;122(5):1849-68. 117. Sung JY, Park SY, Kim JH, Kang HG, Yoon JH, Na YS, et al. Interferon consensus sequence-binding protein (ICSBP) promotes epithelial-to-mesenchymal transition (EMT)-like phenomena, cell-motility, and invasion via TGF-β signaling in U2OS cells. Cell Death Dis. 2014;5:e1224. 118. Lamora A, Talbot J, Bougras G, Amiaud J, Leduc M, Chesneau J, et al. Overexpression of smad7 blocks primary tumor growth and lung metastasis development in osteosarcoma. Clin Cancer Res. 2014;20(19):5097-112. 119. Liu XH, Liu ZL, Sun M, Liu J, Wang ZX, De W. The long non-coding RNA HOTAIR indicates a poor prognosis and promotes metastasis in non-small cell lung cancer. BMC Cancer. 2013;13:464. 120. Lv XB, Lian GY, Wang HR, Song E, Yao H, Wang MH. Long noncoding RNA HOTAIR is a prognostic marker for esophageal squamous cell carcinoma progression and survival. PLoS One. 2013;8(5):e63516. 121. Hajjari M, Behmanesh M, Sadeghizadeh M, Zeinoddini M. Up-regulation of HOTAIR long non-coding RNA in human gastric adenocarcinoma tissues. Med Oncol. 2013;30(3):670. 122. Lu L, Zhu G, Zhang C, Deng Q, Katsaros D, Mayne ST, et al. Association of large noncoding RNA HOTAIR expression and its downstream intergenic CpG island methylation with survival in breast cancer. Breast Cancer Res Treat. 2012;136(3):875-83. 123. Milhem MM, Knutson T, Yang S, Zhu D, Wang X, Leslie KK, et al. Correlation of MTDH/AEG-1 and HOTAIR Expression with Metastasis and Response to Treatment in Sarcoma Patients. J Cancer Sci Ther. 2011;S5(4). 124. Ishibashi M, Kogo R, Shibata K, Sawada G, Takahashi Y, Kurashige J, et al. Clinical significance of the expression of long non-coding RNA HOTAIR in primary hepatocellular carcinoma. Oncol Rep. 2013;29(3):946-50. 125. Kogo R, Shimamura T, Mimori K, Kawahara K, Imoto S, Sudo T, et al. Long noncoding RNA HOTAIR regulates polycomb-dependent chromatin modification and is associated with poor prognosis in colorectal cancers. Cancer Res. 2011;71(20):6320-6. 126. Guffanti A, Iacono M, Pelucchi P, Kim N, Soldà G, Croft LJ, et al. A transcriptional sketch of a primary human breast cancer by 454 deep sequencing. BMC Genomics. 2009;10:163. 127. Yamada K, Kano J, Tsunoda H, Yoshikawa H, Okubo C, Ishiyama T, et al. Phenotypic characterization of endometrial stromal sarcoma of the uterus. Cancer Sci. 2006;97(2):10612. 77 128. Luo JH, Ren B, Keryanov S, Tseng GC, Rao UN, Monga SP, et al. Transcriptomic and genomic analysis of human hepatocellular carcinomas and hepatoblastomas. Hepatology. 2006;44(4):1012-24. 129. Zheng HT, Shi DB, Wang YW, Li XX, Xu Y, Tripathi P, et al. High expression of lncRNA MALAT1 suggests a biomarker of poor prognosis in colorectal cancer. Int J Clin Exp Pathol. 2014;7(6):3174-81. 130. Schmidt LH, Spieker T, Koschmieder S, Schäffers S, Humberg J, Jungen D, et al. The long noncoding MALAT-1 RNA indicates a poor prognosis in non-small cell lung cancer and induces migration and tumor growth. J Thorac Oncol. 2011;6(12):1984-92. 131. Lai MC, Yang Z, Zhou L, Zhu QQ, Xie HY, Zhang F, et al. Long non-coding RNA MALAT-1 overexpression predicts tumor recurrence of hepatocellular carcinoma after liver transplantation. Med Oncol. 2012;29(3):1810-6. 132. Pang EJ, Yang R, Fu XB, Liu YF. Overexpression of long non-coding RNA MALAT1 is correlated with clinical progression and unfavorable prognosis in pancreatic cancer. Tumour Biol. 2014 (epub ahead of print). 78 Anexo A – Ficha de coleta de dados O Identificação 1 1 1 2 Nome 2 2 2 3 Registro Hospitalar 3 3 3 4 Sexo 4 4 1- Feminino; 2- Masculino 4 DD/MM/AAAA 5 1- SUS; 2- Convênios; 3- Outros____________ 6 1- Branco; 2- Negro; 3- Pardo; 4- Amarelo; 99- Ignorado 7 DESCREVER 8 DESCREVER 9 5 Data de nascimento 5 5 6 Categoria 6 6 7 Raça 7 7 8 Naturalidade – Cidade 8 8 9 Naturalidade – UF 9 9 10 11 12 13 14 15 Estado Civil 1 1- Solteiro; 2- Casado / União estável; 3- Separado; 4- Viúvo; 99- Ignorado 1 Atividade profissional DESCREVER 1 Departamento que o atende na nossa instituição DESCREVER 1 Origem do paciente 1- Clínica particular; 2- Rede pública; 3- Por conta própria; 99- Ignorado 1 Data do primeiro atendimento em nosso serviço DD/MM/AAAA 1 Data do diagnóstico DD/MM/AAAA HEMOGRAMA HCB 1 HB 16 1 10 1 11 1 12 1 13 1 14 1 15 1 16 1 HT 1 17 17 1 Leucócitos 1 18 18 1 Neutrófilos 1 19 19 2 Plaquetas 2 20 20 TRATAMENTO HCB 2 Revisão diagnóstica 21 2 0- Não; 1- Sim; 99- Ignorado 21 DESCREVER 22 0- Não; 1- Sim; 99- Ignorado 23 0- Não; 1- Sim; 99- Ignorado 24 DD/MM/AAAA 25 DESCREVER 26 2 Revisão AP 22 2 2 Modificação diagnóstica 23 2 2 Biópsia 24 2 2 Data da biópsia 25 2 2 Local do tumor 26 2 79 2 Data em que recebeu o primeiro tratamento 27 2 DD/MM/AAAA 27 0- Não; 1- Sim; 99- Ignorado 28 DESCREVER 29 0- Não; 1- Sim; 99- Ignorado Motivo da interrupção / Não seguimento no protocolo – Quimioterapia neoadjuvante 3 1- Crescimento do tumor após indução; 2- Ulceração / Sangramento do tumor; 3- Dor intratável; 4- Outros ____________ 3Quimioterapia adjuvante 0- Não; 1- Sim; 99- Ignorado 3Protocolo quimioterapia adjuvante DESCREVER 3Mudança protocolo quimioterapia adjuvante 0- Não; 1- Sim; 99- Ignorado Motivo da interrupção / Não seguimento no protocolo – Quimioterapia adjuvante 3 1- Crescimento do tumor após indução; 2- Ulceração / Sangramento do tumor; 3- Dor intratável; 4- Outros ____________ 3Radioterapia 0- Não; 1- Sim; 99- Ignorado 3Cirurgia 0- Não; 1- Sim; 99- Ignorado 3Data da cirurgia DD/MM/AAAA 3Tipo de Cirurgia 1- Urgente; 2- Eletiva; 88- Não se aplica; 99- Ignorado Tipo de ressecção 4 1- Com preservação do membro; 2- Sem preservação do membro (amputação); 88- Não se aplica; 99- Ignorado Se realizou cirurgia, qual resultado? 4 1- Completamente ressecado com margens livres; 2- Completamente ressecado com margens comprometidas; 3- Ressecção incompleta 4Reconstrução 1- Biológica; 2- Mecânica; 88- Não se aplica; 99- Ignorado 4Complicações cirúrgicas 0- Não; 1- Sim; 88- Não se aplica; 99- Ignorado Infecção de ferida operatória 4 0- Não; 1- Sim, precoce (até 30 dias); 2- Sim, tardia; 88- Não se aplica; 99Ignorado 4Eventos tromboembólicos 0- Não; 1- TVP; 2- TEP Deiscência de ferida operatória 4 0- Não; 1- Sim, sem necessidade de reabordagem; 2- Sim, com necessidade de reabordagem; 88- Não se aplica; 99- Ignorado 4Tipo histológico DESCREVER 4Huvos (Aplicavel apenas para Osteossarcoma) 1- Grau 1; 2- Grau 2; 3- Grau 3; 4- Grau 4; 88- Não se aplica; 99- Ignorado 30 2 Quimioterapia neoadjuvante 28 2 2 Protocolo quimioterapia neoadjuvante 29 2 3 Mudança protocolo quimioterapia neoadjuvante 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 3 3 31 3 32 3 33 3 34 3 35 3 36 3 37 3 38 3 39 4 40 4 41 4 42 4 43 4 44 4 45 4 46 4 47 4 48 80 4Estadiamento utilizado 4 49 49 5Metástases 50 5 0- Ausentes; 1- Presentes 50 DD/MM/AAAA 51 0- Não; 1- Sim; 99- Ignorado 52 0- Não; 1- Sim; 99- Ignorado 53 0- Não; 1- Sim; 99- Ignorado 54 0- Não; 1- Sim; 99- Ignorado 55 0- Não; 1- Sim; 99- Ignorado 56 0- Não; 1- Sim; 99- Ignorado 57 DD/MM/AAAA 58 5Data da metástase 51 5 5Local da metástase - Pulmão 52 5 5Local da metástase - Ossos 53 5 5Local da metástase - Medula Óssea 54 5 5Local da metástase - SNC 55 5 5Local da metástase – Outro local 56 5 5Cirurgia de tórax 57 5 5Data da cirurgia de tórax 58 5 5DHL 5 59 59 6Data da última informação / óbito 60 6 DD/MM/AAAA 60 Status 6 61 62 OBS.: 1- Vivo sem doença; 2- Vivo com doença; 3- Óbito por câncer; 4Óbito por outra doença 6Perdeu seguimento (tempo do retorno x 2) 0- Não; 1- Sim; 99- Ignorado 6 61 6 62 81 Anexo B – Carta de aprovação do Comitê de Ética em Pesquisa do Hospital de Câncer de Barretos e do Hospital das Clínicas da Faculdade de Medicina de Ribeirão Preto/Universidade de São Paulo. 82 83 84


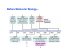
![ORIGEM DA VIDA - AULA ÚNICA [Modo de Compatibilidade] ORIGEM DA VIDA - AULA ÚNICA [Modo de Compatibilidade]](http://s1.doczz.com.br/store/data/000281309_1-c1ffe895897ed723bac75dbc0d94880e-70x70.png)





