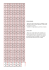INNO-LiPA CFTR17+Tn Update - Biometrix
Transcrição
INNO-LiPA CFTR17+Tn Update - Biometrix
Rev. 01 – Jun/2013
INNO-LiPA CFTR17+Tn Update
Instruções de Uso
USO PRETENDIDO
O INNO-LiPA CFTR17+Tn Update é um ensaio de sondas em linha para
identificação e detecção in vitro de 17 mutações do gene humano Regulador
de Condutância Transmembrana da Fibrose Cística (CFTR), as respectivas
sequências de tipo selvagem, e o polimorfismo CFTR Tn em amostras de
sangue total, swabs bucais e manchas de sangue.
O kit INNO-LiPA CFTR17+Tn Update permite o teste de despiste
multiparamétrico para mutações do gene CFTR e discrimina, pelas mutações
incluídas, entre indivíduos normais saudáveis, transportadores saudáveis, e
pacientes afetados. Este teste também identifica a possível presença de homo
ou heterozigóticos ou a ausência do alelo 5T, que é uma mutação frequente na
doença congênita da ausência bilateral dos vasos deferentes (CBAVD) dos
doentes com penetrância incompleta. Isso permite um aconselhamento
genético mais exato, baseado na classe da mutação do gene da CFTR e da
variante Tn observada, e ajuda a determinar o prognóstico clínico no caso de
doentes com Fibrose Cística.
PRINCÍPIO DO TESTE
Os testes INNO-LiPA CFTR são baseados no princípio da hibridização reversa. O
material com DNA amplificado e biotinilado é quimicamente desnaturado, e as
cadeias separadas são hibridizadas com sondas de oligonucleotídeos
específicas imobilizadas como linhas paralelas em tiras de membranas de
suporte. A isto se segue um passo de lavagem rigorosa para remover qualquer
material amplificado incompatível. Após a lavagem rigorosa, é adicionado o
conjugado de estreptavidina com fosfatase alcalina que se vai ligar aos
híbridos com biotina previamente formados. A incubação com a solução
substrato contendo um cromógeno origina um precipitado castanho-púrpuro. A
reação é interrompida com uma etapa de lavagem, e a matriz de reatividade
das sondas é registrada.
O kit INNO-LiPA CFTR17+Tn Update, é acompanhado por um kit de
amplificação multiplex (Amplificação CFTR), contendo duas soluções com
iniciadores ("primers") e tampões de amplificação, que está disponível para a
preparação padronizada de material amplificado com biotina. Esta
amplificação é baseada na reação das cadeias pela polimerase (PCR).
Os produtos amplificados são subsequentemente hibridizados usando uma tira
onde estão fixadas 36 linhas de sondas de DNA de sequências específicas,
1
Rev. 01 – Jun/2013
uma linha controle e duas linhas marcadoras (figura 1, Ver Resultados).
INNO-LiPA CFTR17+Tn Update: passos envolvidos
Passo 1 - Duas reações de amplificação multiplex:
Éxon/íntron 4, íntron 5, éxon 10, íntron 10/éxon 11, íntron 12, íntron 16,
éxon 20 e éxon 21 do gene CFTR
Éxon 2, íntron 1/íntron 3, éxon 3, éxon 7, íntron 8/éxon 9, éxon 13,
íntron 14b, éxon 17a, íntron 17a/ éxon 17b, éxon 19 e íntron 19 do gene
CFTR
Passo 2 - Hibridização e lavagem rigorosa das 36 sondas imobilizadas na tira
INNO-LiPA CFTR17+Tn Update
Passo 3 - Desenvolvimento da cor
Passo 4 - Interpretação da matriz de reatividade das sondas
REAGENTES
Descrição, modo de preparo e condições de armazenamento recomendado
-
-
-
Reagentes nos frascos originais não abertos e conservados a 2 - 8°C,
são estáveis até o fim do prazo de validade do kit. Não congelar os
reagentes. Não usar os reagentes além do prazo de validade.
O kit deve ser conservado isolado de qualquer fonte contaminante de
DNA, especialmente produtos amplificados.
Todos os reagentes devem ser postos à temperatura ambiente (20 25°C), aproximadamente 60 minutos antes de serem usados e devem
voltar à geladeira imediatamente depois de usados.
Alterações do aspecto físico dos componentes do kit podem indicar
instabilidade ou deterioração.
Para minimizar a possibilidade de enrolamento das tiras antes de serem
utilizadas, recomenda-se conservar o tubo na horizontal.
2
Rev. 01 – Jun/2013
REAGENTES FORNECIDOS
COMPONENTE
QUANTIDADE
Tiras
1 x 20
Solução de
desnaturação
1 x 1 mL
Solução de
Hibridização
1 x 80 mL
Solução de
Lavagem
Rigorosa
1 x 200 mL
Conjugado 100x
1 x 0.8 mL
Diluente do
Conjugado
1 x 80 mL
Substrato
BCIP/NBT 100x
1 x 0.8 mL
Tampão
Substrato
1 x 180 mL
Solução de
Lavagem 5x
1 x 80 mL
Suportes de
Incubação
Cartão de
Leitura
Folhas de
relatório
DESCRIÇÃO
Contém 20 tiras INNO-LiPA CFTR17+Tn Update
marcadas com uma linha superior castanha e uma
segunda linha azul por cima da sonda Tn para
opcional tipificação Tn.
Solução alcalina contendo EDTA. Este frasco deve
ser fechado logo após o uso; exposições
prolongadas desta solução ao ar causa rápida
deterioração da força desnaturante.
Tampão SSC, contendo 0.5% SDS, a ser préaquecido à temperatura de, pelo menos, 37°C e
não exceder os 47°C (os cristais devem ser
dissolvidos antes de usar).
Tampão SSC contendo 0.1% SDS, a ser préaquecido à temperatura de, pelo menos, 37°C e
não exceder os 47°C (os cristais devem ser
dissolvidos antes de usar).
Estreptavidina marcada com fosfatase alcalina em
tampão Tris contendo proteínas estabilizadoras e
0.01% MIT/0.098% CAA como conservante, a ser
diluído a 1/100 em Diluente de Conjugado:
preparar 2 mL de solução de trabalho de
Conjugado para cada tira teste + 2 mL em excesso
para procedimento manual. Para Auto-LiPA,
preparar 10 mL em excesso. A solução de trabalho
de Conjugado é estável por 24 horas em
temperatura ambiente (20 - 25°C) conservada no
escuro.
Tampão fosfato contendo NaCl, Triton, proteínas
estabilizadoras e 0.01% MIT/0.1% CAA como
conservante.
BCIP e NBT em DMF, a ser diluído a 1/100 em
Tampão Substrato antes de usar: preparar 2 mL
de solução de trabalho de Substrato para cada tira
teste + 2 mL em excesso para procedimento
manual. Para Auto-LiPA, preparar 10 mL em
excesso. A solução de trabalho de Substrato é
estável por 24 horas em temperatura ambiente
(20 - 25°C) conservada no escuro.
Tampão Tris contendo NaCl, MgCl2 e 0.01% MIT/0.1%
CAA como conservante.
Tampão fosfato contendo NaCl, Triton e 0.05%
MIT/0.5% CAA como conservante, a ser diluído a 1/5
em água destilada antes de usar: preparar
8 mL de solução de lavagem de trabalho por cada tira
teste + 10 mL em excesso para procedimento manual.
Para Auto-LiPA, prepare 12 mL de Solução de trabalho
Rinse por teste a realizar, mais 20 mL em excesso. A
solução de lavagem de trabalho é estável por 2
semanas a 2 - 8°C.
3
-
1
Para identificação das sondas positivas.
2
Para conservar as tiras reveladas.
3
Rev. 01 – Jun/2013
MATERIAIS NECESSÁRIOS, MAS NÃO INCLUSOS
-
Amplificação CFTR.
Banho-maria com plataforma de agitação (80 rpm; com tampa
inclinada; temperatura ajustável a 47°C ± 0.5°C).
Sistema de aspiração.
Termômetro calibrado.
Agitador orbital, recíproco ou plataforma com agitação lateral.
Recomendações
Para agitador orbital:
O diâmetro do movimento circular deve ser igual ou superior a13 mm
A velocidade recomendada para movimento circular de 13 mm é 160 rpm
Para agitador recíproco:
A velocidade recomendada de vai vem é de 80 movimentos por minuto
Para plataforma com agitação lateral:
O ângulo de agitação não deve exceder 13° para evitar salpicar
líquidos
A velocidade recomendada é de 50 rpm
-
Vortex ou equivalente.
Provetas graduadas (10, 25, 50, e 100 mL).
Àgua destilada ou deionizada.
Luvas descartáveis.
Pontas descartáveis estéreis para pipetas (de preferência com algodão).
Pinça para manusear as tiras.
Pipetas ajustáveis para dispensar 1 - 20 µL, 20 - 200 µL, e 200 - 1000 µL.
Multipipeta dispensadora (Eppendorf, opcional).
Relógio, 2 horas (± 1 minuto).
SEGURANÇA E MEIO AMBIENTE
Tóxico! (T) R61-20/21-36, S53-9-20-23-36/37/39-45-60
Contém N,N-dimetilformamida: SUBS BCIP/NBT 100x.
Irritante! (Xi) R43, S23-24-37-60
Contém 2-cloroacetamida: RINSE SOLN 5x, SUBS BUF, e CONJ DIL.
Corrosiva! (C) R34, S20-23-26-36/37/39-45-60
Contém hidróxido de sódio: DENAT SOLN.
4
Rev. 01 – Jun/2013
R34
R20/21
R36
R43
R61
S9
S20
S23
S24
S26
S36/37/39
S37
S45
S53
utilização.
S60
-
Provoca queimaduras.
Nocivo por inalação e em contato com a pele.
Irritante para os olhos.
Pode causar sensibilização por contato com a pele.
Risco durante a gravidez com efeitos adversos na descendência.
Manter o recipiente num local bem ventilado.
Não comer nem beber durante a utilização.
Não inalar vapor/aerosol.
Evitar o contato com a pele.
Em caso de contato com os olhos, lavar imediata e
abundantemente com água e consultar um especialista.
Usar vestuário de proteção, luvas e equipamento protetor para os
olhos/face adequados.
Usar luvas adequadas.
Em caso de acidente ou indisposição, consulte um médico de
imediato (mostre o rótulo sempre que possível).
Evitar a exposição - obter instruções específicas antes da
Este produto e o seu recipiente devem ser eliminados como
resíduos perigosos.
As amostras devem ser processadas como potencialmente infecciosas.
Por esse motivo, todos os componentes sanguíneos e materiais
biológicos devem ser considerados como potencialmente infecciosos e
manipulados como tal. Somente pessoal treinado deve ser permitido a
executar o procedimento do teste. Todos os componentes sanguíneos e
materiais biológicos devem ser destruídos de acordo com os
procedimentos de segurança estabelecidos.
Autoclavar pelo menos durante 15 minutos a 121°C.
Incinerar o material descartável.
Misturar os líquidos de esgoto com hipoclorito de sódio de modos a que
a concentração seja ± 1% em hipoclorito de sódio. Deixar em contato
toda a noite antes de despejar.
ATENÇÃO: Neutralizar o líquido de descarte que contém ácido antes de
adicionar o hipoclorito de sódio.
-
É necessário o uso de equipamento de proteção pessoal: luvas e óculos
de segurança quando manipular agentes perigosos ou infecciosos.
O material de descarte deve ser manipulado de acordo com as linhas de
orientação da instituição. Todas as regulamentações estatais e locais
sobre meio ambiente devem ser respeitadas.
5
Rev. 01 – Jun/2013
AMOSTRAS
Uma vez que o teste INNO-LiPA CFTR17+Tn Update utiliza como amostra o
material DNA amplificado com biotina, o kit de amplificação, Amplificação
CFTR, está disponível e faz parte do mesmo para amplificações multiplex das
respectivas 8 e 11 regiões do gene CFTR (solução primer 1 e solução primer
2).
PROCEDIMENTO DE TESTE LiPA
O procedimento LiPA padrão descreve o teste de uma tira em uma cavidade.
No entanto, um estudo de equivalência mostrou que o INNO-LiPA CFTR17+Tn
Update pode ser testado na mesma cavidade do INNO-LiPA CFTR19. Por
conseguinte, deve adicionar 10 µL de produtos de amplificação individuais a
10 µL de solução de desnaturação, enquanto mantém todos os outros
parâmetros de protocolo iguais.
PROCEDIMENTOS DE EXECUÇÃO
Manipulação das tiras
-
-
As tiras foram desenvolvidas para serem usadas uma única vez!
Não tocar nas tiras com as mãos; usar luvas limpas.
Usar um lápis para identificação das tiras. Não usar esferográficas,
marcadores, etc. Escrever a identificação acima da linha de marcação
das tiras.
Colocar as tiras teste nas tinas de incubação com as membranas
revestidas viradas para cima (lado com a marca).
Durante os diferentes passos de incubação, as tiras teste devem
permanecer sempre na mesma tina de incubação.
As tiras não usadas ou reveladas devem ser conservadas ao abrigo da
luz intensa e do calor.
Deixar as tiras teste reveladas secar completamente antes de
interpretar, cobrir e guardar.
As tiras teste reveladas devem ser conservadas de preferência no
escuro e à temperatura ambiente (20 - 25°C).
Não reutilizar os suportes de incubação.
Instruções para incubação manual
-
A incubação a 47°C ± 0.5°C durante a hibridização e lavagem rigorosa,
são os passos mais críticos para evitar falsos positivos (temperatura
muito baixa) ou falsos negativos/sinais muito fracos (temperatura muito
alta). Um banho-maria com agitador e tampa inclinada permitem um
bom controle das variações da temperatura. É necessário um controle
6
Rev. 01 – Jun/2013
rigoroso da temperatura com um termômetro calibrado.
Fechar sempre a tampa do banho durante as incubações de modos a
evitar falsos sinais positivos.
Não usar um agitador de ar quente para a hibridização e lavagem
rigorosa.
Para a hibridização e lavagem rigorosa, os suportes de incubação
devem estar colocados na plataforma agitadora do banho. Ajustar o
nível da água entre 1/3 e 1/2 da altura dos suportes. Verificar que os
suportes não flutuam na água. A água deve estar em contato direto com
os suportes.
Os passos de incubação para o desenvolvimento da cor devem ser feitos
entre 20 e 25°C. Se a temperatura estiver abaixo de 20°C, podem obterse resultados fracos. Se a temperatura estiver acima dos 25°C, pode
obter-se um sinal de fundo e/ou sinais falsos positivos.
A amplitude de agitação gerada pelos agitadores, seja durante a
hibridização ou durante o desenvolvimento da cor, é critica, de modo a
alcançar o máximo de sensibilidade e coloração homogênea. As
superfícies das tiras devem estar completamente submersas. A
amplitude deve ser tão alta quanto possível de modo a permitir que
tanto os líquidos como as tiras teste se movam para frente e para trás
no respectivo suporte. Contudo, respingos de liquido para as bordas do
suporte deve ser evitado! Isto pode levar a contaminação cruzada e a
resultados inválidos.
Não cobrir o suporte. Durante as incubações de hibridização e lavagem
rigorosa no banho, os suportes devem ficar destapados. Cobrir os
suportes com os selantes de microplacas pode levar a contaminação
cruzada.
-
-
-
-
Instruções para aspirar e dispensar manualmente soluções das tinas de
incubação
-
O líquido deve ser aspirado da tina com uma pipeta, de preferência
ligada a um aspirador de vácuo. O suporte deve manter-se com um
ângulo que permita o escoamento do líquido para uma das
extremidades.
Adicionar 2 mL da solução apropriada a cada tina e seguir o
procedimento do teste. O uso de uma Multipipeta (Eppendorf) facilita
esta operação.
Repetir este passo tantas vezes quantas as indicadas no procedimento
do teste.
-
-
NOTA:
Não deixar secar as tiras durante dois passos.
Verificar que não danifica a superfície das tiras quando faz a aspiração.
Aspirar o líquido no topo da tira, acima da linha de marcação.
Verificar que todas as tiras são lavadas convenientemente por
7
Rev. 01 – Jun/2013
completa submersão na solução.
Adaptar a velocidade do agitador se necessário.
OBSERVAÇÕES E PRECAUÇÕES
-
-
Só para uso de profissionais.
Para evitar contaminação de DNA, recomenda-se a máxima separação
física entre as etapas pré e de pós-amplificação: salas separadas,
pipetas e outros materiais de laboratório separados, separar batas e
luvas (e seu estoque) são as precauções mínimas para uma boa prática
de laboratório.
Evitar voltar da sala de pós-amplificação para a de pré-amplificação.
Recomenda-se o uso de material descartável autoclavado.
Não reutilizar material descartável.
Usar uma ponta estéril nova por alíquota de amostra.
Não usar reagentes com o prazo validade expirado.
Evitar a contaminação microbiana dos reagentes.
PROCEDIMENTO PARA TESTE MANUAL LiPA
NOTA:
-
Durante os diferentes passos de incubação, as tiras teste devem
permanecer nas mesmas tinas.
Antes da incubação, verificar a temperatura (47°C ± 0.5°C) do banho usando
um termômetro calibrado, e ajustar a temperatura se necessário antes de
colocar o suporte com as tinas de incubação. Fechar sempre a tampa.
Amostras
1. Produto CFTR amplificado (usar 10 µL de produto amplificado com a solução
de primer PS parte 1 + 10 µL de produto amplificado com PS parte 2). Para
preparação das amostras consultar as instruções do kit de Amplificação CFTR.
NOTA:
Verificar que são adicionados exatamente 10 µL de ambas partes
da amostra amplificada. Demasiada ou muito pouca amostra
pode levar a um resultado de tipagem errado.
2. Branco amplificado controle (controles negativos; usar 10 µL de ambos
controles negativos (ver instruções da Amplificação CFTR).
8
Rev. 01 – Jun/2013
Desnaturação e hibridização
1. Aquecer o banho com agitação a 47°C ± 0.5°C. Verificar a temperatura
usando um termômetro calibrado, e ajustar a temperatura se necessário. Não
exceder as temperaturas indicadas. Pré-aquecer a Solução de
Hibridização e a Solução de lavagem rigorosa num banho-maria pelo menos a
37°C não excedendo os 47°C. Agitar antes de usar. Todos os cristais devem
ser dissolvidos.
2. Com luvas, retirar o número necessário de tiras de INNO-LiPA CFTR17+Tn
Update do tubo (uma tira por cada amostra). Incluir uma tira para o controle
negativo. Identificar as amostras escrevendo o número a lápis acima da linha
de marcação da tira.
3. Colocar as canaletas de incubação (uma canaleta por cada amostra a
testar) no suporte.
4. Pipetar 10 µL de Solução de Desnaturação no canto superior de cada tina.
NOTA:
Fechar o frasco imediatamente após usar.
5. Adicionar 10 µL do produto amplificado com a solução de primer PS parte 1
e misturar cuidadosamente com a Solução de Desnaturação pipetando para
cima e para baixo. Utilizar sempre pontas estéreis.
6. Adicionar Imediatamente 10 µL do produto amplificado com a solução de
primer PS parte 2 e misturar cuidadosamente com a Solução de Desnaturação
pipetando para cima e para baixo. Utilizar sempre ponteiras estéreis. Deixar
processar a desnaturação durante 5 minutos a 20 - 25°C.
7. Agitar a Solução de Hibridização pré-aquecida e dispensar gentilmente 2 mL
em cada tina com produto desnaturado amplificado. Misturar com agitação
suave. Ter em atenção não contaminar as canaletas vizinhas durante a
pipetagem.
8. Colocar Imediatamente as tiras em cada canaleta com a linha de marcação
da membrana voltada para cima. As tiras devem ficar completamente
submergidas na solução.
NOTA:
Usar luvas descartáveis e pinça.
9. Colocar o suporte no banho a 47°C ± 0.5°C com agitação (80 rpm; ver
instruções de incubação manual), fechar a tampa e incubar 90 minutos.
9
Rev. 01 – Jun/2013
NOTA:
Evitar salpicar água do banho para dentro das canaletas. Ajustar
o nível de água entre 1/3 e 1/2 da altura da canaleta. Para
prevenir movimentos laterais do suporte, imobilizar com auxílio
de dois pesos.
Lavagem rigorosa
1. Após hibridização, retirar o suporte do banho.
2. Segurar o suporte com uma leve inclinação ao aspirar o líquido das
canaletas com uma pipeta, de preferência ligada ao vácuo. Adicionar 2 mL de
Solução de Lavagem Rigorosa em cada tina e lavar agitando ligeiramente o
suporte (10 a 20 segundos) a 20 - 25°C. Aspirar a solução de cada tina. Repetir
esta lavagem mais uma vez (ver também as instruções para mudar
manualmente as soluções das tinas).
3. Finalmente, aspirar a solução e incubar cada tira em 2 mL de Solução de
Lavagem Rigorosa pré-aquecida no banho a 47°C ± 0.5°C com agitação
durante 30 minutos. Fechar a tampa do banho.
Antes da incubação, verificar a temperatura da água do banho usando um
termômetro calibrado, e ajustar a temperatura se necessário. Fechar sempre a
tampa.
NOTA:
Diluir as Soluções de Lavagem e de Conjugado concentradas
durante a lavagem rigorosa. Ver Reagentes.
Desenvolvimento da cor
Todas as incubações subsequentes são efetuadas a 20 - 25°C num agitador.
Durante as incubações, a solução e as tiras devem mover-se para trás e para
frente nas canaletas para obter uma coloração homogênea.
1. Lavar cada tira duas vezes durante 1 minuto usando 2 mL de Solução de
Lavagem diluída (ver Reagentes e Instruções para mudar soluções das tinas).
2. Adicionar 2 mL de Solução de Conjugado diluída (ver Reagentes) a cada tina
e incubar durante 30 minutos sob agitação.
NOTA:
Diluir a Solução de Substrato BCIP/NBT cerca de 10 minutos antes
do fim da incubação do Conjugado. Ver Reagentes.
3. Lavar cada tira duas vezes durante 1 minuto usando 2 mL de Solução de
Lavagem diluída e lavar uma vez mais usando 2 mL de Tampão Substrato.
10
Rev. 01 – Jun/2013
4. Adicionar 2 mL de Solução de Substrato diluída (ver Reagentes) a cada e
incubar 30 minutos sob agitação.
5. Parar o desenvolvimento da cor lavando as tiras duas vezes com 2 mL de
água destilada sob agitação durante pelo menos 3 minutos.
6. Com auxílio de uma pinça, retirar as tiras das canaletas e colocar em papel
absorvente. Deixar secar as tiras completamente antes de ler os resultados.
Conservar as tiras reveladas e secas no escuro.
PROCEDIMENTO PARA TESTE AUTOMATIZADO LiPA
Os procedimentos de teste automatizados estão disponíveis. Contate a
Biometrix para obter mais informações.
RESULTADOS
Leitura
A Figura 1 ilustra a posição das diferentes sondas de oligonucleotídeos na tira
do INNO-LiPA CFTR17+Tn Update. Uma linha é considerada positiva quando
aparece uma banda definida de cor púrpura/castanha no final da execução do
teste.
Figura 1: Localização da linha de marcação superior (linha castanha), a segunda linha de marcação
(linha azul) em cima das sondas Tn, a linha do control e do conjugado e as 36 sondas de DNA de
sequência específica sobre a tira INNO-LiPA CFTR17+Tn Update.
11
Rev. 01 – Jun/2013
Validação
-
-
-
-
-
Incluir uma amostra de controle de amplificação negativa, cada vez que
executar o teste. Tal como qualquer procedimento de um novo teste, a
inclusão adicional de controles positivos e negativos deve ser
considerada até que se adquira um elevado grau de confiança que
permita a execução correta do teste. Sugere-se a utilização de DNA de
referência que possa ser usado para validação em como as condições
próprias do teste foram estabelecidas.
A primeira linha da tira do INNO-LiPA CFTR17+Tn Update é uma linha de
marcação (linha castanha). Esta linha permite uma orientação correta
da tira.
A linha seguinte controla a adição da reatividade das Soluções de
Conjugado e de Substrato durante o procedimento de detecção. Esta
linha deve ser alinhada com a linha de controle do Conjugado do cartão
plástico de leitura. Esta linha tem que ser sempre positiva e deve
apresentar aproximadamente a mesma intensidade em cada tira de
uma mesma série.
O resultado de cada amostra de controle de amplificação negativa não
deve apresentar sinal em qualquer das linhas da tira, exceto para a
linha de controle do Conjugado.
A intensidade das cores das sondas numa tira pode diferir de uma linha
para outra.
Interpretação dos resultados
-
-
-
Primeiro verifique a positividade da linha controle (primeira linha após a
linha de marcação superior) de modo a validar cada tira individualmente
(ver Validação).
A linha de marca da tira (castanha) deve ser alinhada em cima com a
linha de marca do cartão plástico de leitura.
A tira INNO-LiPA CFTR17+Tn Update contem 16 sondas tipo-selvagem
(Wild-types), 17 sondas tipo-mutante (Mutant.-types) e 3 sondas Tn .
W.2183AA→G=W.2184delA é a sequência tipo-selvagem para
2183AA→G e 2184delA.
Identificar todas as sondas que são positivas na tira INNO-LiPA
CFTR17+Tn Update.
Interpretação das sondas W.-type e M.-type: A presença de uma linha
visível significa que uma ou outra das sequências W.-type ou M.-type
estão presentes num ou em ambos alelos. Indivíduos, homozigotos para
certo alelo W.-type apresentam um sinal positivo só para a
correspondente sonda W.-type, e não para a correspondente sonda M.type. Indivíduos, homozigotos para certo alelo M.-type apresentam um
sinal positivo só para a correspondente sonda M.-type, e não para a
correspondente
sonda
W.-type.
Indivíduos,
heterozigotos
ou
heterozigotos compostos para determinada(s) mutação (ões)
apresentam sinal positivo para ambas as sondas correspondentes W.- e
12
Rev. 01 – Jun/2013
-
-
-
M.-type.
Interpretação das sondas Tn (opcional): A presença de uma linha visível
significa que a correspondente sequência Tn está presente em um ou
em ambos os alelos. Indivíduos, homozigotos para certo alelo Tn
apresentam um sinal positivo só para a sonda Tn correspondente.
Quando é detectado R117H, o resultado T5 ou T7 também deve ser
estabelecido.
R117H pode ser encontrado na forma cis com T5 ou T7. R117H-T5 é
considerado como um leve alelo causador de um complexo CF;
enquanto a forma R117H-T7 é considerada (com maior probabilidade)
como relacionada com uma mutação CFTR.
Quando detectado na forma heterozigota composta com uma mutação
causadora de CF, ou possivelmente até mesmo em homozigose R117HT5 geralmente resulta em CF pancreática, enquanto R117H-T7 pode
resultar em uma forma leve de CF, azoospermia obstrutiva, ou em
nenhuma doença.
(Castellani C, Cuppens H, Macek JR M et al. Consensus on the use and
interpretation of cystic fibrosis mutation analysis in clinical practice. J Cyst
Fibros 2008; 7:179-196).
(Dequeker E, Stuhrmann M, Morris MA et al. Best practice guidelines for
molecular genetic diagnosis of cystic fibrosis and CFTR-related disorders –
updated European recommendations. Eur j Hum Genet 2008; 1-15).
Aparentes perfis de reação aberrantes
-
-
-
-
Como W.2183AA→G é a sequência selvagem (W.-type) de 2183AA→G
assim como para 2184delA, um homozigótico 2183AA→G/2183AA→G,
um homozigótico 2184delA/2184delA, ou um heterozigoto composto
2183AA→G/2184delA a amostra do paciente não apresenta sinal com a
sonda W.2183AA→G.
Como W.711+5G→A (presente na tira do INNO-LiPA CFTR17+Tn Update)
é a sequência selvagem para 711+5G→A assim como para 711+1G→T
(presente
na tira do INNO-LiPA
CFTR19), um homozigoto
711+1G→T/711+1G→T, um homozigoto 711+5G→A/711+5G→A, ou um
heterozigoto composto 711+1G→T/711+5G→A a amostra do paciente
não apresenta sinal com a sonda W.711+5G→A.
Para uma amostra de um paciente homozigoto CFTRdele2,3
(21kb)/CFTRdele2,3 (21kb), não se obtém sinal com sequências
selvagens do éxon3 (W.E60X, W.G85E, e W.394delTT) do INNO-LiPA
CFTR17+Tn Update.
As outras seguintes mutações raras da CFTR foram avaliadas por reação
cruzada e não foram detectadas pelo teste INNO-LiPA CFTR17+Tn
Update: 2184insA (éxon 13), 2789+3delG (íntron 14b), R347H (éxon 7),
R117C (éxon 4), 621+2T→C (íntron 4), e 711+3A→G (íntron 5).
13
Rev. 01 – Jun/2013
-
-
Não é obtido sinal com a sonda W.R1162X quando o paciente é
homozigoto para o polimorfismo 3617G->T.
Foi observada uma reatividade cruzada da sonda 9T com o raro e
identificado (Kobler D, Modi H, Goldman B. Identification of an 11T allele
in the polypyrimidine tract of íntron 8 of the CFTR gene. Genet Med
2006;8:125-8) alelo 11T.
Foi observada reatividade cruzada para a mutação R117P com a sonda
R117H.
LIMITES DO PROCEDIMENTO
-
-
-
-
O uso deste produto deve ser limitado só a pessoas treinadas em
técnicas de amplificação e hibridização.
Só uma boa prática de laboratório e uma execução cuidadosa dos
procedimentos especificados, permitem uma hibridização específica e
uma tipagem correta do DNA alvo.
Uma não interpretação do resultado pode ocorrer devido à presença de
polimorfismos da CFTR ou outras mutações raras de CFTR na região das
nossas sondas ou na parte 3' dos nossos primers.
Para uma amostra heterozigota composta, o teste LiPA não permite
revelar se as mutações estão localizadas em um ou dois cromossomos.
Contudo amostras de DNA dos pais podem também ser testadas com o
INNO-LiPA CFTR17+Tn Update para reduzir o risco de uma interpretação
errada.
É possível que outras mutações estejam presentes, mas que não estão
incluídas no kit e, como tal, não são detectadas.
O DNA muito puro com uma concentração próxima do limite superior do
intervalo de aplicação pode conduzir a reações não específicas.
DESEMPENHO DO TESTE INNO-LiPA CFTR19 e INNO-LiPA CFTR17+Tn Update
-
-
-
O desempenho do INNO-LiPA CFTR19 e INNO-LiPA CFTR17+Tn Update foi
avaliado externamente em dois laboratórios Belgas de genética
humana. Ao todo, foram analisadas 101 amostras anônimas.
As hibridizações foram efetuadas manualmente em um centro e usando
o Auto-LiPA em outro. Os resultados foram interpretados visualmente.
Foi documentado um genótipo para cada amostra combinando os dados
obtidos das duas tiras.
Os cálculos da sensibilidade e exatidão de diagnóstico só tiveram em
conta as mutações incluídas nas tiras do INNO-LiPA CFTR19 e INNO-LiPA
CFTR17+Tn Update. Uma amostra foi excluída dos cálculos, pois o
resultado do ensaio de referência revelou uma mutação que não pôde
ser detectada com o INNO-LiPA CFTR19 e INNO-LiPA CFTR17+Tn Update
(711+3A→G) (ver discussão abaixo).
14
Rev. 01 – Jun/2013
-
O teste de sensibilidade do ensaio foi calculado como o número de
resultados positivos (amplificação com sucesso e resultado interpretável
no LiPA) sobre o número total de amostras testadas.
O teste de sensibilidade nesta avaliação foi de 100% (100/100; 95% CI [96%;
100%]).
-
-
A exatidão do Diagnóstico foi calculada como o número de genótipos
corretamente identificados comparado com o resultado obtido com um
ou mais ensaios alternativos (INNO-LiPA, Innogenetics®; Elucigene,
Orchid; sequenciamento; métodos caseiros). Foi obtida uma total
concordância com o resultado de referência, resultando uma exatidão
de diagnóstico de 100% (100/100; 95% CI [96%; 100%]). Os
polimorfismos Tn foram comparáveis para 51 amostras, todos os
resultados foram idênticos.
Mutações presentes na tira, mas não disponíveis nos centros, assim
como um grande número de outras mutações, foram avaliadas
internamente. Todas as amostras foram corretamente genotipadas.
Além disso, foram avaliadas 13 amostras de zaragatoas bucais e 20
amostras de manchas de sangue seco de dadores de sangue.
Em cada caso, os resultados obtidos com os swabs bucais e as manchas de
sangue seco foram idênticos aos resultados das amostras de sangue dos
mesmos indivíduos. A reatividade global dos resultados das tiras obtidos com
as manchas de sangue seco e os swabs bucais foi menos intensa, quando
comparada com as reações das sondas obtidas com as amostras de sangue
total.
Reatividades das sondas
-
-
O desenvolvimento dos ensaios teve o objetivo de evitar reações
cruzadas com outras mutações ou polimorfismos no gene CFTR. As
seguintes mutações/polimorfismos, encontradas nos centros, não
apresentaram qualquer reação cruzada com as sondas presentes nas
tiras: R117C, R347H, F508C. Dados internos provam a ausência de
reação cruzada com 2184insA. Para uma amostra heterozigota, foi
observada uma reação cruzada para a mutação 1898+1G→C com a
sonda 1898+1G→A.
Reatividade não específica de bandas mutantes foi observada em três
amostras em um centro, mas não voltaram a aparecer na repetição do
teste. De acordo com o investigador, a reatividade não especifica foi
devida a um contato prematuro entre a tira e o amplicon/solução de
desnaturação. O outro centro também identificou reatividade não
específica para várias amostras, as quais, contudo, não interferiram com
a interpretação. Observações internas e dos dados dos centros,
sugerem que deve ter tido como causa o uso de DNA muito puro e a
uma concentração próxima do limite superior do intervalo de aplicação.
15
Rev. 01 – Jun/2013
-
A seção Interpretação dos resultados do folheto informativo menciona a
possibilidade de as sondas tipo selvagem poderem não dar reatividade.
Exemplos encontrados na avaliação externa foram a mutação
homozigota 711+1G→T (sonda tipo selvagem 711+5G→A/711+1G→T),
e F508del/I507del (sonda tipo selvagem F508del/I507del). Devido à
especificidade das sondas mutantes, podem ser esperadas reatividades
muito fracas ou não reatividade com amostras contendo mutações não
presentes na tira, mas localizadas na região das nossas sondas.
Por exemplo, a mutação homozigota 711+3A→G não apresentou reatividade
com qualquer outra sonda tipo selvagem (como seria de esperar com uma
amostra mutante homozigótica) ou sondas mutante para 711+1G→T ou
711+5G→A. A mutação homozigota 2184insA não apresentou reatividade com
a sonda mutante 2184delA (foi observada uma reatividade muito fraca com
sonda tipo selvagem 2184delA). Onde não se obtiverem resultados
interpretáveis para sequências tipo selvagem ou mutante na posição 711 e
2184 respectivamente, o clínico é alertado para posteriores investigações
destas amostras nestas posições específicas.
Precisão
Testes internos incluíram 8 amostras, analisadas em triplicado, em 3 séries
diferentes, com 3 lotes de tiras diferentes, tanto manualmente como no Auto LiPA, por 3 pessoas diferentes. Não se observou variação significativa e a
interpretação do genótipo foi idêntica em todos os casos. Um painel de
proficiência, constituído por 5 amostras com reatividades conhecidas, foi
fornecido aos dois centros externos e também analisados internamente. Os
resultados dos três laboratórios apresentaram uma completa concordância.
IDENTIFICAÇÃO DO DISTRIBUIDOR
Biometrix Diagnóstica Ltda.
Estrada da Graciosa, 1081 - Curitiba – PR - CEP: 82840-360
Tel.: (41) 2108-5250
Fax: (41) 2108-5252
DDG: 0800-7260504
E-mail: [email protected]
Website: www.biometrix.com.br
CNPJ: 06.145.976/0001-39
16
Rev. 01 – Jun/2013
INFORMAÇÕES DO FABRICANTE
INNOGENETICS N.V.
Technologiepark 6
9052 Gent - Belgium
Tel.: +32-9 329 13 29
REGISTRO ANVISA
80433150012
RESPONSÁVEL TÉCNICA
Edna Cristina Kurokawa Guimarães Ferreira
CRQ/PR: 09302336
17






 REAGENTES FORNECIDOS
COMPONENTE
REAGENTES FORNECIDOS
COMPONENTE